Mer om søking i sekvenser
|
|
|
- Roy Dalen
- 7 år siden
- Visninger:
Transkript
1 Mer om søking i sekvenser agens forelesning orelesning IN335/ sept 27 Ole Christian Lingjærde Gruppen for bioinformatikk Institutt for Informatikk, UiO Kort oppsummering fra sist Globale sammenstillinger med ulineære scorefunksjoner Optimale lokale sammenstillinger (Smith-aterman) Heuristiske lokale sammenstillinger (BLS) Når er en sammenstilling signifikant? 2 Repetisjon: dynamisk programmering Vi så sist gang på Needleman-unsch algoritmen for optimal global sammenstilling av to sekvenser Repetisjon: dynamisk programmering Vi kunne dermed fylle inn tabellen under med optimale scoreverdier rad for rad (eller kolonne for kolonne): Med en lineær scorefunksjon S() fant vi følgende rekurrensrelasjoner for optimal score B(i,j) mellom :i og y :j : B(,) = Bi (,) = S(, ) + + S( i, ) = ig B(, j) = S(, y ) + + S(, y ) = jg j 2 B(,) B(,) B(2,) y B(,) B(,) B(2,) y 2 B(,2) B(,2) B(2,2) y 3 B(,3) B(,3) B(2,3) y 4 B(,4) B(,4) B(2,4) Bi (, j ) + S( i, yj) B(, i j) = ma B( i, j) + S( i, ) i, j Bi (, j ) + S(, yj ) B(3,) B(4,) B(3,) B(4,) B(3,2) B(4,2) B(3,3) B(4,3) B(3,4) B(4,4) 4
2 Repetisjon: dynamisk programmering Optimering med ulineær scorefunksjon Ved å markere avhengighetsforholdet mellom optimale verdier (hvilke som er basert på hvilke) med piler, kan vi etterpå lett konstruere en optimal vei og tilhørende optimale sammenstilling: Eksempel: S( a, yb) = S(, y) + BLOSUM 62( a, b) gopen, y ender ikke med S( a, y) = S(, y) getend, y ender med a blir: B(,) = Bi (,) = gopen ( i ) getend B(, j) = gopen ( j ) getend Bi (, j ) + BLOSUM62( i, yj) gopen hvis B( i, j) B( i 2, j) Bi (, j) = ma Bi (, j) getend hvis Bi (, j) Bi ( 2, j) gopen hvis B( i, j ) Bi (, j 2) Bi (, j ) getend hvis B( i, j ) B( i, j 2) 6 Eksempel Eksempel S(a,a) = S(a,b) = -3 for a b UIineær gapstraff med g open = 5 og g etend = 2 = y = S(a,a) = S(a,b) = -3 for a b UIineær gapstraff med g open = 5 og g etend = 2 = y =
3 Eksempel S(a,a) = S(a,b) = -3 for a b UIineær gapstraff med g open = 5 og g etend = 2 = y = Eksempel S(a,a) = S(a,b) = -3 for a b UIineær gapstraff med g open = 5 og g etend = 2 = y = Eksempel Lokal parvis sammenstilling S(a,a) = S(a,b) = -3 for a b UIineær gapstraff med g open = 5 og g etend = 2 = y = raceback: Needleman-unsch algoritmen kan lett modifiseres til å finne beste lokale sammenstilling av to sekvenser og y ette gir Smith-aterman algoritmen inner da generelt en sammenstilling av to delsekvenser i:j og y r:s for passende i j og r s Samme prinsipp (regneregler) som før, men vi ser om det er mulig å oppnå enda høyere score ved å utelate symboler i begynnelsen av eller slutten av sekvensene
4 Lokal parvis sammenstilling: ide Rekurrensformler nta at vi er ute etter en lokal sammenstilling med positiv score Utelat sluttsymboler i og y som bidrar negativt til scoren I praksis gjør vi dette ved å starte traceback i den ruten som har størst verdi Utelat startsymboler i og y som bidrar negativt til scoren I praksis gjør vi dette ved å sette inn i tabellen når optimal score blir negativ avslutte traceback før vi kommer til en Utregning av B(i,j)-verdier for lokal parvis sammenstilling med lineær scorefunksjon: B(,) = Bi (,) = i> B(, j) = j > Bi (, j ) + S( i, yj) Bi (, j) = ma i, j > Bi (, j) + S( i, ) Bi (, j ) + S(, yj ) 3 4 Eksempel på lokal sammenstilling S(a,a) = S(a,b) = - for a b Gapstraff g = - = y = Eksempel på lokal sammenstilling S(a,a) = S(a,b) = - for a b Gapstraff g = - = y = 5 6
5 Eksempel på lokal sammenstilling S(a,a) = S(a,b) = - for a b Gapstraff g = - = y = Eksempel på lokal sammenstilling S(a,a) = S(a,b) = - for a b Gapstraff g = - = y = Eksempel på lokal sammenstilling S(a,a) = S(a,b) = - for a b Gapstraff g = - = y = Eksempel på lokal sammenstilling S(a,a) = S(a,b) = - for a b Gapstraff g = - = y =
6 Eksempel på lokal sammenstilling Implementasjon: datastruktur S(a,a) = S(a,b) = - for a b Gapstraff g = - = y = raceback: 2 2 Lokal sammenstilling - Score 2 2 or å implementere Needleman-unsch eller Smith-aterman trenger du en datastruktur for å holde på B(i,j)-verdiene i sammenstillingstabellen (i=,,m; j=,,n) pilene i sammenstillingstabellen Noen muligheter: o m n tabeller: en tabell B for å holde på B(i,j)-verdiene en tabell P for å markere pilretninger (feks = ingen pil, = venstre, 2 = diagonalt, 3 = oppover) En m n 2 - tabell: C(i,j,) = B(i,j) og C(i,j,2) = P(i,j) i notasjonen ovenfor 2 22 Implementasjon: algoritme (Needleman-unsch) lgoritme-kompleksitet lgoritme (innfyllingsfase): Sett B(,) = 2 inn B(i,), i=,,n, og B(,j), j=,,m 3 or i = :n or j = :m Beregn B(i,j) Sett P(i,j) til "venstre", "oppover" eller "diag" basert på resultatet ovenfor nta vi skal sammenstille to sekvenser av lengde m og n Minne: O(nm) Et fast antall tabeller (en eller to) med n*m plasser : konstant * nm Et fast antall hjelpevariable : konstant id: O(nm) Må regne ut B(i,j) og P(i,j) for n*m ruter i tabellen : konstant * nm Må foreta traceback : konstant * (n+m) lgoritme (traceback-fase): Sett i = n og j = m 2 Hvis P(i,j) er "venstre": sammenstill "-" med y[j]; j--; "oppover": sammenstill [i] med "-"; i--; "diag" : sammenstill [i] med y[j]; i--; j--; 3 Hvis i > eller j > : gå til 2 Ellers: stopp 23 nta feks at vi bruker 8 bytes minne pr plass i tabellen (svarende til to int-variable eller en int-variabel og en float-variabel i Java) Sammenstilling av to N-sekvenser av lengde krever * * 8 = 8 Mb minne lengde krever * * 8 = 8 Gb minne 24
7 Optimalisering av minnebruk nta først at vi skal finne beste globale sammenstilling En optimal vei vil som oftest ligge nokså nær hoveddiagonalen (fordi gap straffes) Vi begrenser derfor søkerommet til et bånd rundt linjen som forbinder startruten og sluttruten i tabellen: Hvis m n: Minne: O(Kn) id: O(Kn) K = båndbredden Kun de B(i,j)-verdiene som ligger innenfor båndet beregnes, og ved trace-back går vi aldri utenfor båndet Merk: vi kan risikere å gå glipp av den optimale sammenstillingen (hvis den går utenfor båndet) 25 Optimalisering av minnebruk - 2 nta nå at vi skal finne beste lokale sammenstilling a er startruten og sluttruten for optimal vei i utgangspunktet ukjent inn først ruten B(i,j) med høyest score ette kan gjøres med minne O(n) og i tid O(mn) hvis vi bare lagrer raden vi holder på å fylle inn og raden ovenfor Resultat: Ruten med maksimal score 26 Optimalisering av minnebruk - 3 Lokal sammenstilling med BLS En optimal vei fra den markerte ruten vil som oftest ligge nær den diagonal linjen bakover fra den optimale ruten (fordi gap straffes) efiner et bånd rundt den diagonale linjen Beregn alle B(i,j)-verdier innenfor båndet, og gjør traceback fra den optimale ruten BLS = Basic Local lignment Search ool Omfatter flere nært beslektede metoder: NCBI-BLS (NCBI = National Center for Biotechnology Information) U-BLS (U = ashington University) Hvis m n: Vi ser her på NCBI-BLS (den opprinnelige BLS), og kaller den bare for BLS i fortsettelsen Minne: O(Kn) id: O(mn) K = båndbredden BLS ble til i 989 Målet var å lage et heuristisk søkeprogram som veldig raskt kunne finne rimelig opplagte lokale similariteter mellom sekvenser 27 BLS viste seg imidlertid også å være god til å finne fjernt beslektede sekvenser 28
8 Naboskapstabellen BLS benytter en tabell som angir for hver trimer S alle de trimerer som har høy similaritet med S, definert ved at de sammenstilt med S gir en score Slike trimerer utgjør naboskapet til S orekomsttabellen BLS benytter også en tabell som angir for hver trimer hvor den forekommer i databasen C YY YYY C YY YYY Mer generelt kan trimerer erstattes med -merer ypiske valg er = 3 for proteinsekvenser og = for N-sekvenser Videre kan vi feks ha 29 score = BLOSUM62 og = rimer C YYY orekomster i databasen 3 BLS-algoritmen: hovedtrinnene nta at en søkesekvens Q er gitt: Velg en trimer S i sekvensen Q 2 Velg en S' i naboskapet til S 3 Velg en forekomst av S' la oss si i sekvensen Q' - i databasen 4 Sammenstill S i sekvensen Q med S' i sekvensen Q' Utvid sammenstillingen i begge retninger langs Q og Q', inntil en av følgende inntreffer: BLS-algoritmen: hovedtrinnene forts Kun de beste HSP'ene beholdes (de med score over en gitt terskel) Hver av HSP'ene undersøkes nærmere ved å finne en (tilnærmet) optimal lokal sammenstilling med gap i dette området Beregn E-verdi og endel andre verdier som skal rapporteres til brukeren sammen med den lokale sammenstillingen kkumulert score < ma(akkumulert score) ERSKEL kkumulert score < Enden av en av sekvensene nås 4 Sammenstillingen vi nå har kalles High-scoring Segment Pair (HSP) Prosessen gjentas for alle valg av S, S' og forekomster av S' 3 32
9 BLS: valgmuligheter BLS: valg av type søk ype søk (feks N mot protein) atabase som det skal søkes i Organisme som det skal søkes i ormat på søkesekvens ype sammenstilling (med/uten gap) iltrering av lavkompleksitetsområder Scoringsskjema ormat på BLS-rapporten 33 Program BLSN MEGBLS BLSP PSI-BLS PHI-BLS BLS BLSN BLS atabase Nukleotid Protein Protein Nukleotid (oversettes til protein) Nukleotid (oversettes til protein) Søkesekvens Nukleotid Protein Nukleotid (oversettes til protein) Protein Nukleotid (oversettes til protein) Eksempel på bruk Gitt en N-sekvens, finn relaterte N-sekvenser i samme organisme eller i andre organismer Gitt en proteinsekvens, finn beslektede proteiner Gitt en N-sekvens, avgjør om den svarer til kjent protein Gitt en proteinsekvens, finn posisjonen til tilhørende gen Gitt en N-sekvens som potensielt koder for protein inn relaterte Nsekvenser i samme organisme eller andre organismer (mer følsom enn BLSN) 34 BLS: valg av database (eksempler) Peptidsekvens-databaser nr ll non-redundant GenBank CS translations+pb+swissprot+pir+pr swissprot Last major release of the SISS-PRO protein sequence database (no updates) yeast Yeast (Saccharomyces cerevisiae) genomic CS translations ecoli Escherichia coli genomic CS translations pdb Sequences derived from the 3-dimensional structure from PB alu ranslations of select lu repeats from REPBSE Nukleotidsekvens-databaser nr ll GenBank+EMBL+BJ+PB sequences () dbest atabase of GenBank+EMBL+BJ sequences from ES ivisions dbsts atabase of GenBank+EMBL+BJ sequences from SS ivisions htgs Unfinished High hroughput Genomic Sequences yeast Yeast (Saccharomyces cerevisiae) genomic nucleotide sequences E coli Escherichia coli genomic nucleotide sequences pdb Sequences derived from the 3-dimensional structure from PB mito atabase of mitochondrial sequences alu Select lu repeats from REPBSE 35 BLS: valg av format på søkesekvens Sekvens i S-format (se neste foil) GI-nummer (GenBank Identifier) Løpenummer som tildeles sekvenser etterhvert som de blir registrert i GenBank Oppdateringer av sekvensen får nytt GI-nummer ccession # Entydig identifikator som tildeles en sekvensrecord (= en sekvens + tilhørende informasjon) første gang denne blir registrert Senere oppdateringer av en record fører ikke til endring av, eller nye, accession # 36
10 S format IUPC-koder (IUPC = International Union of Pure and pplied Chemistry) ørst en kommentarlinje som starter med > og som gir en kort beskrivelse av dataene, deretter dataene på IUPC-format (se neste foil) Linjene skal helst ha maksimalt 8 tegn denin Cytosin Guanin C G lanin Cystein sparginsyre Glutaminsyre la Cys sp Glu C E Eksempel (N): hymin Uracil U enylalanin Glycin Phe Gly G >B263 acc=b263 descr=homo sapiens mrn for CGGCCGCCCCCGGCCCCGCGCGCGCCCCGGGGCCCGGCC CCGCGCCCGCCCCGGGGGGGCCCCCCGGCCGGCGCGGCGCC GGGCGGCGGGGGCGCCCCGCCCCGCGGGG GGGGCCCCCGGCCGGCCGGGCCCCCGGGGGGCCGGGGG Eksempel (protein): Purin (G ) Pyrimidin ( C) Keto (G ) mino ( C) G eller C eller G, eller C R Y K M S B Histidin Isoleucin Lysin Leucin Methionin sparagin Prolin His Ile Lys Leu Met sn Pro H I K L M N P >gi pir 496 chitinase (EC 324) YPLKLPPSSCVGHKLMNVVVVEQVREYPLKLPPSSCVGHKLMNVVVV EQVREYPLYPLKLPPSSCVGHKLMNVVVVEQVREYPLKLPPYPLK LPPSSCVGHKLMNVVVVEQVREYPLKLPPSSCVGHKLMNVVVVEQVR EYPLYPLKLPPSSCVGHKLMNVVVVEQVREYPLKLPP 37 G, eller, C eller, C, G eller NB: store og små bokstaver kan normalt brukes om hverandre H N Glutamin rginin Serin hreonin Valin ryptofan yrosin Gln rg Ser hr Val rp yr Q R S V Y 38 BLS: valg av scoringsskjema BLS homepage (wwwncbinlmnihgov/blast) Scoringsmatrise BLOSUM45 BLOSUM62 BLOSUM8 PM3 PM7 Gapstraff Kan velge mellom diverse affine gapstraffer Velg søkeprogram 39 4
11 Eksempel: protein blast Eksempel : søkestreng = gi Her oppgis søkesekvensen Valg av database Valg av søkeprogram Start søk 4 42 BLS-rapporten (innledning) BLS-rapporten (grafisk oversikt) Skal refereres når man publiserer noe basert på NCBI-BLS Resultat-identifikator (RI) Info om databasen Info om søkesekvensen 43 44
12 BLS-rapporten (liste over treff i databasen) Ett av treffene i databasen Bit score / raw score E-verdi Her kan vi velge å se nærmere på de enkelte treffene HSP HSP = High-scoring Segment Pair BLS versus dynamisk programmering Egenskaper ved BLS: Er to sekvenser relaterte? nta at vi har en lokal sammenstilling: Vesentlig raskere enn dynamisk programmering Utforsker ikke hele spekteret av mulige sammenstillinger, i motsetning til dynamisk programmering inner ikke nødvendigvis alle lokale similariteter som oppfyller kriteriet for å bli rapportert inner statistisk signifikante sammenstillinger selv om de ikke er optimale, i motsetning til dynamisk programmering Slike sammenstillinger kan være av stor biologisk interesse : 2 3 N y: y y 2 y 3 y N Sammenstillingen kan være funnet ved: Smith-aterman BLS En annen sammenstillingsalgoritme Vi antar for enkelhets skyld at sammenstillingen er uten gap Hvordan avgjør vi om sekvensene er relaterte? 47 48
13 Mulige framgangsmåter Hva er mest trolig: relatert eller ikke? Sammenlign sannsynligheten ovenfor med tilsvarende sannsynlighet beregnet under antagelse av at sekvensene er relaterte (og med en viss evolusjonær avstand) Hva er mest trolig? Log-likelihood ratio et er essensielt dette vi gjør når vi regner ut BLOSUM-scoren til en sammenstilling: husk at scoren mellom aminosyre i og aminosyre j er BLOSUM(i,j) = log [q ij / (p i *p j )] I BLS rapporteres denne verdien som raw score (eller bare score) Litt notasjon S( j, k) : score for sammenstilling av aminosyre j og aminosyre k Si () i : akkumulert score Si () = S ( j, y j ) j= p k : a priori sannsynligheten for at en vilkårlig i er aminosyre k : a priori sannsynligheten for at en vilkårlig y i er aminosyre k p k 2 i N y y 2 y i y N 2 Hvor trolig er resultatet hvis de ikke er relaterte? Under antagelse av at det er en tilfeldig sammenstilling av to sekvenser, hvor sannsynlig er det at vi skulle observere en så høy score som obs? ette er prinsippet for Karlin-ltschul testen i BLS 49 S(, y ) 2 2 i Si () = S ( j, y j ) j= 5 Urelaterte sekvenser or urelaterte sekvenser vil vi ha "typiske verdiintervaller" for: andelen par ( i,y i ) med identiske aminosyrer (dvs i =y i ) lengden av lengste sammenhengende segment av slike par lengden av lengste ekskursjon fra et stigepunkt Vi kan så sjekke om den gitte sammenstillingen er i samsvar med dette 5 Urelaterte sekvenser forts nta at sekvensene er urelaterte (i henhold til tidligere modell): sannsynligheten for aminosyreparet (j,k) : p p j forventet score : forventet akkumulert score : i i 2 2 E[ S( i)] = E[ S(, y )] = E[ S(, y )] = i p p S( j, k) Merk at k 2 2 E[ S(, y )] pps ( j, k) = antar at S er valgt slik i i j k j= k= at denne blir negativ aminosyre j aminosyre k i i i i j k j= j= j= k= ES [ ()] > ES [ (2)] > > ESN [ ( )] S(i) har "negativ drift" 52
14 Normalisert score Motivasjon for definisjonen av lambda Ulike scorefunksjoner (scorematriser) S(j,k) kan operere på ulike skalaer il hver scorefunksjon S(j,k) kan vi definere en normalisert score S '( j, k) = λs( j, k) λ > hvor er en skaleringsfaktor Ideen er at disse normaliserte scorene er sammenliknbare for ulike scoringsfunksjoner λ Scaleringsfaktoren bestemmes ved å løse likningen 2 2 j= k= j S( j, k) k = ppe λ et finnes ulike måter å definere scorefunksjonen S(j,k) på, men vi kan nesten alltid tenke på S(j,k) som et estimat på lod-scoren, dvs q jk S( j, k) log (tilnærmet) pp Vi definerer λ slik at j k Etter en enkel omskriving får vi q = p p e λ jk j k Vi summerer over alle j og k og får: j q jk λ S( j, k) = log (tilnærmet) pp k S( j, k) λs( j, k) ppe j k qjk j= k= j= k= = = E-verdi og P-verdi Søk mot en hel database La s være scoren til en HSP et kan vises at når sekvensene er urelaterte, så vil forventet antall ekskursjoner av høyde s eller mer være tilnærmet E = k Ne λs (E-verdi, eller Epect-verdi) Hittil har vi tatt utgangspunkt i en lokal sammenstilling uten gap av to sekvenser som begge har lengde N I praksis er situasjonen ofte en annen: Vi har en søkesekvens som består av m aminosyrer Vi søker mot en database som totalt består av n aminosyrer en konstant (kan beregnes) ølgende resultat kan også vises å holde tilnærmet: Prob( S s) = e E lengden av sekvensene som sammenstilles (P-verdi) en statistiske analysen er mye mer komplisert i dette tilfellet (og er ennå ikke fullstendig avklart) ølgende resultat er etablert: Når søkesekvensen og databasesekvensen er urelaterte, vil forventet antall ekskursjoner av høyde s eller mer være tilnærmet E = kmne λ s 55 for en passende konstant k (som er kjent) 56
15 olking av P-verdier olking av E-verdier Lave P-verdier svarer til høy signifikans Eksempel: Lave E-verdier svarer til høy signifikans Retningslinjer: P < - : eksakt match P < -5 : nær identiske P < - : nært beslektet P < - : fjernt beslektet (eller ikke beslektet) ellers : ikke signifikant E < 2 : trolig beslektet E < : kan være beslektet ellers : ingen indikasjon på beslektet 57 58
Gensøk. Oppsummering. Typer av sammenstillinger. Sammenstilling av sekvenser. To prinsipper for søking etter gener i DNA:
 Oppsummering Gensøk Oppsummeringen som gis her omfatter bare de temaer som er forelest av Ole Christian, og er ikke ment å være komplett. I korte trekk gjelder for denne delen av pensum som for de øvrige:
Oppsummering Gensøk Oppsummeringen som gis her omfatter bare de temaer som er forelest av Ole Christian, og er ikke ment å være komplett. I korte trekk gjelder for denne delen av pensum som for de øvrige:
BLAST. Blast. Noen mulige sammenstilling av CHAEFAP og CAETP. Evolusjonær basis for sekvenssammenstilling. Sekvenssammenstilling og statistikken brukt
 Blast BLAST Sekvenssammenstilling og statistikken brukt Finner best mulig sammenstilling(er), evt. finner veldig gode sammenstillinger. Kan teoretisk unngå å finne beste sammenstilling. Avgjør om sammenstillingen
Blast BLAST Sekvenssammenstilling og statistikken brukt Finner best mulig sammenstilling(er), evt. finner veldig gode sammenstillinger. Kan teoretisk unngå å finne beste sammenstilling. Avgjør om sammenstillingen
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF2300 Grunnkurs i bioinformatikk Eksamensdag : Tirsdag 15. juni 2004 Tid for eksamen : 09.00 12.00 Oppgavesettet er på : 13
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF2300 Grunnkurs i bioinformatikk Eksamensdag : Tirsdag 15. juni 2004 Tid for eksamen : 09.00 12.00 Oppgavesettet er på : 13
Søk i sekvenser. Sekvenssøk. Sammenstillinger av sekvenser. Global og lokal sammenstilling. Global sammenstilling: Lokal sammenstilling:
 øk i sekvenser ekvenssøk Forelesning NF3350/4350 9. sept 2007 Ole Christian ingjærde Gruppen for bioinformatikk nstitutt for nformatikk, UiO Mål:identifisere hvilke sekvenser i en sekvensdatabase som er
øk i sekvenser ekvenssøk Forelesning NF3350/4350 9. sept 2007 Ole Christian ingjærde Gruppen for bioinformatikk nstitutt for nformatikk, UiO Mål:identifisere hvilke sekvenser i en sekvensdatabase som er
UNIVERSITETET I OSLO
 Kandidatnummer: BOKMÅL UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF3350/INF4350 Grunnkurs i bioinformatikk Eksamensdag : Tirsdag 5. desember 2006 Tid for eksamen : 15.30
Kandidatnummer: BOKMÅL UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF3350/INF4350 Grunnkurs i bioinformatikk Eksamensdag : Tirsdag 5. desember 2006 Tid for eksamen : 15.30
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF2300 Grunnkurs i bioinformatikk Eksamensdag : Mandag 6. juni 2005 Tid for eksamen : 09.00 12.00 Oppgavesettet er på : xx
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF2300 Grunnkurs i bioinformatikk Eksamensdag : Mandag 6. juni 2005 Tid for eksamen : 09.00 12.00 Oppgavesettet er på : xx
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2017 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2017, forelesning
INF2220 - Algoritmer og datastrukturer HØSTEN 2017 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2017, forelesning
IN Algoritmer og datastrukturer
 IN010 - Algoritmer og datastrukturer HØSTEN 018 Institutt for informatikk, Universitetet i Oslo Forelesning 6: Grafer III Ingrid Chieh Yu (Ifi, UiO) IN010 0.10.018 1 / 0 Dagens plan: Dybde-først søk Biconnectivity
IN010 - Algoritmer og datastrukturer HØSTEN 018 Institutt for informatikk, Universitetet i Oslo Forelesning 6: Grafer III Ingrid Chieh Yu (Ifi, UiO) IN010 0.10.018 1 / 0 Dagens plan: Dybde-først søk Biconnectivity
Generelle Tips. INF Algoritmer og datastrukturer. Åpen og Lukket Hashing. Hashfunksjoner. Du blir bedømt etter hva du viser at du kan
 Generelle Tips INF2220 - lgoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo Du blir bedømt etter hva du viser at du kan Du må begrunne svar Du må ikke skrive av bøker
Generelle Tips INF2220 - lgoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo Du blir bedømt etter hva du viser at du kan Du må begrunne svar Du må ikke skrive av bøker
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2016 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2016, forelesning
INF2220 - Algoritmer og datastrukturer HØSTEN 2016 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2016, forelesning
Søking i strenger. Prefiks-søking Naiv algoritme Knuth-Morris-Pratt-algoritmen Suffiks-søking Boyer-Moore-algoritmen Hash-basert Karp-Rabin-algoritmen
 Søking i strenger Vanlige søkealgoritmer (on-line-søk) Prefiks-søking Naiv algoritme Knuth-Morris-Pratt-algoritmen Suffiks-søking Boyer-Moore-algoritmen Hash-basert Karp-Rabin-algoritmen Indeksering av
Søking i strenger Vanlige søkealgoritmer (on-line-søk) Prefiks-søking Naiv algoritme Knuth-Morris-Pratt-algoritmen Suffiks-søking Boyer-Moore-algoritmen Hash-basert Karp-Rabin-algoritmen Indeksering av
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 11: Huffman-koding & Dynamisk programmering (Ifi, UiO) INF2220 H2015, forelesning 11 1 / 32 Dagens
INF2220 - Algoritmer og datastrukturer Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 11: Huffman-koding & Dynamisk programmering (Ifi, UiO) INF2220 H2015, forelesning 11 1 / 32 Dagens
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 13: Eksamensgjennomgang Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 13 1 /
INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 13: Eksamensgjennomgang Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 13 1 /
Turingmaskiner.
 Turingmaskiner http://www.youtube.com/watch?v=e3kelemwfhy http://www.youtube.com/watch?v=cyw2ewoo6c4 Søking i strenger Vanlige søkealgoritmer (on-line-søk) Prefiks-søking Naiv algoritme Knuth-Morris-Pratt-algoritmen
Turingmaskiner http://www.youtube.com/watch?v=e3kelemwfhy http://www.youtube.com/watch?v=cyw2ewoo6c4 Søking i strenger Vanlige søkealgoritmer (on-line-søk) Prefiks-søking Naiv algoritme Knuth-Morris-Pratt-algoritmen
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 5: Prioritetskø og Heap Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 5 1 /
INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 5: Prioritetskø og Heap Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 5 1 /
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Kandidatnr Eksamen i INF1000 Grunnkurs i objektorientert programmering Eksamensdag: Onsdag 1. desember 2010 Tid for eksamen: 14.00 18.00
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Kandidatnr Eksamen i INF1000 Grunnkurs i objektorientert programmering Eksamensdag: Onsdag 1. desember 2010 Tid for eksamen: 14.00 18.00
Dagens plan. INF Algoritmer og datastrukturer. Koding av tegn. Huffman-koding
 Grafer Dagens plan INF2220 - Algoritmer og datastrukturer HØSTEN 2007 Institutt for informatikk, Universitetet i Oslo Avsluttende om grådige algoritmer (kap. 10.1.2) Dynamisk programmering Floyds algoritme
Grafer Dagens plan INF2220 - Algoritmer og datastrukturer HØSTEN 2007 Institutt for informatikk, Universitetet i Oslo Avsluttende om grådige algoritmer (kap. 10.1.2) Dynamisk programmering Floyds algoritme
IN Algoritmer og datastrukturer
 IN2010 - Algoritmer og datastrukturer HØSTEN 2018 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 3: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2010 H2018, forelesning
IN2010 - Algoritmer og datastrukturer HØSTEN 2018 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 3: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2010 H2018, forelesning
Mer om gensøk. Kjapp oppsummering fra sist gang. Motif eller tilfeldig DNA forts. Motif eller tilfeldig DNA? Forelesning INF3350/
 Mer om gensøk Kjapp oppsummering fra sist gang Forelesning INF3350/4350 12. sept 2007 Ole Christian Lingjærde Gruppen for bioinformatikk Institutt for Informatikk, UiO To prinsipper for søking etter gener
Mer om gensøk Kjapp oppsummering fra sist gang Forelesning INF3350/4350 12. sept 2007 Ole Christian Lingjærde Gruppen for bioinformatikk Institutt for Informatikk, UiO To prinsipper for søking etter gener
Testsituasjon Resultat Kommentar. Fungerer som det skal!
 Test- rapport Testsituasjon Resultat Kommentar Test av PHP-variablene. Sjekke om de er riktig deklarert, og om de kommer med fra form til database Alle variablene som skal leses fra konfigurasjonssiden,
Test- rapport Testsituasjon Resultat Kommentar Test av PHP-variablene. Sjekke om de er riktig deklarert, og om de kommer med fra form til database Alle variablene som skal leses fra konfigurasjonssiden,
INF 4130 / / Dagens foiler hovedsakelig laget av Petter Kristiansen Foreleser Stein Krogdahl Obliger:
 INF 4130 / 9135 29/8-2012 Dagens foiler hovedsakelig laget av Petter Kristiansen Foreleser Stein Krogdahl Obliger: Tre stykker, som må godkjennes. Frister: 21. sept, 26. okt, 16. nov Andre, «nærliggende»
INF 4130 / 9135 29/8-2012 Dagens foiler hovedsakelig laget av Petter Kristiansen Foreleser Stein Krogdahl Obliger: Tre stykker, som må godkjennes. Frister: 21. sept, 26. okt, 16. nov Andre, «nærliggende»
Stein Krogdahl, Dino Karabeg, Petter Kristiansen. Kenneth A. Berman and Jerome L. Paul.
 Stein Krogdahl, Dino Karabeg, Petter Kristiansen steinkr at ifi.uio.no dino at ifi.uio.no pettkr at ifi.uio.no INF 4130 / 9135 Algoritmer: Design og effektivitet Algorithms: Sequential Parallel and Distributed
Stein Krogdahl, Dino Karabeg, Petter Kristiansen steinkr at ifi.uio.no dino at ifi.uio.no pettkr at ifi.uio.no INF 4130 / 9135 Algoritmer: Design og effektivitet Algorithms: Sequential Parallel and Distributed
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2015 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2015, forelesning
INF2220 - Algoritmer og datastrukturer HØSTEN 2015 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2015, forelesning
Maps og Hashing. INF Algoritmer og datastrukturer. Map - ADT. Map vs Array
 Maps og Hashing INF0 - Algoritmer og datastrukturer HØSTEN 00 Institutt for informatikk, Universitetet i Oslo INF0, forelesning : Maps og Hashing Map - Abstrakt Data Type Hash-funksjoner hashcode Kollisjonshåndtering
Maps og Hashing INF0 - Algoritmer og datastrukturer HØSTEN 00 Institutt for informatikk, Universitetet i Oslo INF0, forelesning : Maps og Hashing Map - Abstrakt Data Type Hash-funksjoner hashcode Kollisjonshåndtering
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2015 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2015, forelesning
INF2220 - Algoritmer og datastrukturer HØSTEN 2015 Ingrid Chieh Yu Institutt for informatikk, Universitetet i Oslo Forelesning 4: Prioritetskø og Heap Ingrid Chieh Yu (Ifi, UiO) INF2220 H2015, forelesning
UNIVERSITETET I OSLO
 Bokmål UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF2300 Grunnkurs i bioinformatikk Eksamensdag : Mandag 6. juni 2005 Tid for eksamen : 09.00 12.00 Oppgavesettet er på
Bokmål UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i : INF2300 Grunnkurs i bioinformatikk Eksamensdag : Mandag 6. juni 2005 Tid for eksamen : 09.00 12.00 Oppgavesettet er på
6 Determinanter TMA4110 høsten 2018
 6 Determinanter TMA4110 høsten 2018 En matrise inneholder mange tall og dermed mye informasjon så mye at det kan være litt overveldende Vi kan kondensere ned all informasjonen i en kvadratisk matrise til
6 Determinanter TMA4110 høsten 2018 En matrise inneholder mange tall og dermed mye informasjon så mye at det kan være litt overveldende Vi kan kondensere ned all informasjonen i en kvadratisk matrise til
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 13: Dynamisk programmering (Ifi, UiO) INF2220 H2017, forelesning 13 1 / 30 Dagens plan Dynamisk
INF2220 - Algoritmer og datastrukturer Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 13: Dynamisk programmering (Ifi, UiO) INF2220 H2017, forelesning 13 1 / 30 Dagens plan Dynamisk
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 3: Maps og Hashing Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 3 1 / 25 Maps
INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 3: Maps og Hashing Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 3 1 / 25 Maps
INF1010 notat: Binærsøking og quicksort
 INF1010 notat: Binærsøking og quicksort Ragnhild Kobro Runde Februar 2004 I dette notatet skal vi ta for oss ytterligere to eksempler der rekursjon har en naturlig anvendelse, nemlig binærsøking og quicksort.
INF1010 notat: Binærsøking og quicksort Ragnhild Kobro Runde Februar 2004 I dette notatet skal vi ta for oss ytterligere to eksempler der rekursjon har en naturlig anvendelse, nemlig binærsøking og quicksort.
MAT1030 Diskret Matematikk
 MAT1030 Diskret Matematikk Forelesning 29: Kompleksitetsteori Roger Antonsen Institutt for informatikk, Universitetet i Oslo 13. mai 2009 (Sist oppdatert: 2009-05-17 22:38) Forelesning 29: Kompleksitetsteori
MAT1030 Diskret Matematikk Forelesning 29: Kompleksitetsteori Roger Antonsen Institutt for informatikk, Universitetet i Oslo 13. mai 2009 (Sist oppdatert: 2009-05-17 22:38) Forelesning 29: Kompleksitetsteori
GRAFER. Korteste vei i en vektet graf uten negative kanter. Korteste vei, en-til-alle, for: Minimale spenntrær
 IN Algoritmer og datastrukturer GRAER IN Algoritmer og datastrukturer Dagens plan: orteste vei, en-til-alle, for: ektet rettet graf uten negative kanter (apittel 9..) (Dijkstras algoritme) ektet rettet
IN Algoritmer og datastrukturer GRAER IN Algoritmer og datastrukturer Dagens plan: orteste vei, en-til-alle, for: ektet rettet graf uten negative kanter (apittel 9..) (Dijkstras algoritme) ektet rettet
Backtracking som løsningsmetode
 Backtracking Backtracking som løsningsmetode Backtracking brukes til å løse problemer der løsningene kan beskrives som en sekvens med steg eller valg Kan enten finne én løsning eller alle løsninger Bygger
Backtracking Backtracking som løsningsmetode Backtracking brukes til å løse problemer der løsningene kan beskrives som en sekvens med steg eller valg Kan enten finne én løsning eller alle løsninger Bygger
Forelesning 29: Kompleksitetsteori
 MAT1030 Diskret Matematikk Forelesning 29: Kompleksitetsteori Roger Antonsen Institutt for informatikk, Universitetet i Oslo Forelesning 29: Kompleksitetsteori 13. mai 2009 (Sist oppdatert: 2009-05-17
MAT1030 Diskret Matematikk Forelesning 29: Kompleksitetsteori Roger Antonsen Institutt for informatikk, Universitetet i Oslo Forelesning 29: Kompleksitetsteori 13. mai 2009 (Sist oppdatert: 2009-05-17
MAT1030 Plenumsregning 3
 MAT1030 Plenumsregning 3 Ukeoppgaver Mathias Barra - 30. januar 2009 (Sist oppdatert: 2009-02-02 14:26) Plenumsregning 3 Oppgave 2.7 - Horners metode (a) 7216 8 : 7 8+2 58 8+1 465 8+6 3726. Svar: 3726
MAT1030 Plenumsregning 3 Ukeoppgaver Mathias Barra - 30. januar 2009 (Sist oppdatert: 2009-02-02 14:26) Plenumsregning 3 Oppgave 2.7 - Horners metode (a) 7216 8 : 7 8+2 58 8+1 465 8+6 3726. Svar: 3726
Maps og Hashing. INF Algoritmer og datastrukturer. Map - ADT. Map vs Array
 Maps og Hashing INF0 - Algoritmer og datastrukturer HØSTEN 00 Institutt for informatikk, Universitetet i Oslo INF0, forelesning : Maps og Hashing Map - Abstrakt Data Type (kapittel.) Hash-funksjoner (kapittel..)
Maps og Hashing INF0 - Algoritmer og datastrukturer HØSTEN 00 Institutt for informatikk, Universitetet i Oslo INF0, forelesning : Maps og Hashing Map - Abstrakt Data Type (kapittel.) Hash-funksjoner (kapittel..)
MAT1030 Diskret matematikk
 MAT1030 Diskret matematikk Forelesning 30: Kompleksitetsteori Dag Normann Matematisk Institutt, Universitetet i Oslo 14. mai 2008 Informasjon Det er lagt ut program for orakeltjenestene i MAT1030 denne
MAT1030 Diskret matematikk Forelesning 30: Kompleksitetsteori Dag Normann Matematisk Institutt, Universitetet i Oslo 14. mai 2008 Informasjon Det er lagt ut program for orakeltjenestene i MAT1030 denne
Hashing. INF Algoritmer og datastrukturer HASHING. Hashtabeller
 Hashing INF2220 - Algoritmer og datastrukturer HØSTEN 200 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning : Hashing Hashtabeller (kapittel.) Hash-funksjoner (kapittel.2) Kollisjonshåndtering
Hashing INF2220 - Algoritmer og datastrukturer HØSTEN 200 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning : Hashing Hashtabeller (kapittel.) Hash-funksjoner (kapittel.2) Kollisjonshåndtering
Dagens plan: INF Algoritmer og datastrukturer. Eksempel. Binære Relasjoner
 Dagens plan: INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 10: Disjunkte Mengder Definisjon av binær relasjon Definisjon av ekvivalens
Dagens plan: INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 10: Disjunkte Mengder Definisjon av binær relasjon Definisjon av ekvivalens
Korteste vei i en vektet graf uten negative kanter
 Dagens plan: IN - Algoritmer og datastrukturer HØSTEN 7 Institutt for informatikk, Universitetet i Oslo IN, forelesning 7: Grafer II Korteste vei, en-til-alle, for: Vektet rettet graf uten negative kanter
Dagens plan: IN - Algoritmer og datastrukturer HØSTEN 7 Institutt for informatikk, Universitetet i Oslo IN, forelesning 7: Grafer II Korteste vei, en-til-alle, for: Vektet rettet graf uten negative kanter
FYS 3710 Biofysikk og Medisinsk Fysikk, Aminosyrer, Polypeptider, Proteiner
 FYS 3710 Biofysikk og Medisinsk Fysikk, 2016 5 Aminosyrer, Polypeptider, Proteiner Einar Sagstuen, Fysisk institutt, UiO 06.09.2016 1 sp n -hybridisering: for hovedkvantetall N=2 er de fire valensorbitalene
FYS 3710 Biofysikk og Medisinsk Fysikk, 2016 5 Aminosyrer, Polypeptider, Proteiner Einar Sagstuen, Fysisk institutt, UiO 06.09.2016 1 sp n -hybridisering: for hovedkvantetall N=2 er de fire valensorbitalene
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 10: Disjunkte Mengder Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 10 1 / 27
INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo INF2220, forelesning 10: Disjunkte Mengder Bjarne Holen (Ifi, UiO) INF2220 H2009, forelesning 10 1 / 27
MAT1030 Diskret matematikk
 MAT1030 Diskret matematikk Plenumsregning 9: Diverse ukeoppgaver Roger Antonsen Matematisk Institutt, Universitetet i Oslo 10. april 2008 Oppgaver fra forelesningene Oppgave (fra forelesningen 10/3) a)
MAT1030 Diskret matematikk Plenumsregning 9: Diverse ukeoppgaver Roger Antonsen Matematisk Institutt, Universitetet i Oslo 10. april 2008 Oppgaver fra forelesningene Oppgave (fra forelesningen 10/3) a)
INF2220: Forelesning 1. Praktisk informasjon Analyse av algoritmer (kapittel 2) (Binær)trær (kapittel )
 INF2220: Forelesning 1 Praktisk informasjon Analyse av algoritmer (kapittel 2) (Binær)trær (kapittel 4.1-4.3 + 4.6) PRAKTISK INFORMASJON 2 Praktisk informasjon Kursansvarlige Ragnhild Kobro Runde (ragnhilk@ifi.uio.no)
INF2220: Forelesning 1 Praktisk informasjon Analyse av algoritmer (kapittel 2) (Binær)trær (kapittel 4.1-4.3 + 4.6) PRAKTISK INFORMASJON 2 Praktisk informasjon Kursansvarlige Ragnhild Kobro Runde (ragnhilk@ifi.uio.no)
INF Algoritmer og datastrukturer
 INF2220 - Algoritmer og datastrukturer HØSTEN 2016 Institutt for informatikk, Universitetet i Oslo Forelesning 6: Grafer II Ingrid Chieh Yu (Ifi, UiO) INF2220 28.09.2016 1 / 30 Dagens plan: Dijkstra fort.
INF2220 - Algoritmer og datastrukturer HØSTEN 2016 Institutt for informatikk, Universitetet i Oslo Forelesning 6: Grafer II Ingrid Chieh Yu (Ifi, UiO) INF2220 28.09.2016 1 / 30 Dagens plan: Dijkstra fort.
Oppgaver fra forelesningene. MAT1030 Diskret matematikk. Oppgave (fra forelesningen 10/3) Definisjon. Plenumsregning 9: Diverse ukeoppgaver
 Oppgaver fra forelesningene MAT1030 Diskret matematikk Plenumsregning 9: Diverse ukeoppgaver Roger Antonsen Matematisk Institutt, Universitetet i Oslo 10. april 2008 Oppgave (fra forelesningen 10/3) a)
Oppgaver fra forelesningene MAT1030 Diskret matematikk Plenumsregning 9: Diverse ukeoppgaver Roger Antonsen Matematisk Institutt, Universitetet i Oslo 10. april 2008 Oppgave (fra forelesningen 10/3) a)
INF1020 Algoritmer og datastrukturer GRAFER
 GRAFER Dagens plan: Avsluttende om grådige algoritmer Huffman-koding (Kapittel 10.1.2) Dynamisk programmering Floyds algoritme for korteste vei alle-til-alle (Kapittel 10.3.4) Ark 1 av 16 Forelesning 22.11.2004
GRAFER Dagens plan: Avsluttende om grådige algoritmer Huffman-koding (Kapittel 10.1.2) Dynamisk programmering Floyds algoritme for korteste vei alle-til-alle (Kapittel 10.3.4) Ark 1 av 16 Forelesning 22.11.2004
Hashtabeller. Lars Vidar Magnusson Kapittel 11 Direkte adressering Hashtabeller Chaining Åpen-adressering
 Hashtabeller Lars Vidar Magnusson 12.2.2014 Kapittel 11 Direkte adressering Hashtabeller Chaining Åpen-adressering Dictionaries Mange applikasjoner trenger dynamiske sett som bare har dictionary oparsjonene
Hashtabeller Lars Vidar Magnusson 12.2.2014 Kapittel 11 Direkte adressering Hashtabeller Chaining Åpen-adressering Dictionaries Mange applikasjoner trenger dynamiske sett som bare har dictionary oparsjonene
LP. Leksjon 6: Kap. 6: simpleksmetoden i matriseform, og Seksjon 7.1: følsomhetsanalyse
 LP. Leksjon 6: Kap. 6: simpleksmetoden i matriseform, og Seksjon 7.1: følsomhetsanalyse matrisenotasjon simpleksalgoritmen i matrisenotasjon eksempel negativ transponert egenskap: bevis følsomhetsanalyse
LP. Leksjon 6: Kap. 6: simpleksmetoden i matriseform, og Seksjon 7.1: følsomhetsanalyse matrisenotasjon simpleksalgoritmen i matrisenotasjon eksempel negativ transponert egenskap: bevis følsomhetsanalyse
MAT1030 Diskret matematikk
 MAT1030 Diskret matematikk Plenumsregning 3: Ukeoppgaver fra kapittel 2 & 3 Roger Antonsen Matematisk Institutt, Universitetet i Oslo 31. januar 2008 Oppgave 2.7 - Horners metode (a) 7216 8 : 7 8+2 58
MAT1030 Diskret matematikk Plenumsregning 3: Ukeoppgaver fra kapittel 2 & 3 Roger Antonsen Matematisk Institutt, Universitetet i Oslo 31. januar 2008 Oppgave 2.7 - Horners metode (a) 7216 8 : 7 8+2 58
Foreleser: Eivind Coward, kontor 5. etg. Datablokken. coward@ii.uib.no Gruppeleder: Harald Barsnes
 Foreleser: Eivind Coward, kontor 5. etg. Datablokken. coward@ii.uib.no Gruppeleder: Harald Barsnes Forelesninger: tirsdag og fredag 12 14 rom 2104 Øvinger: fredag 10 12 rom 2143 Gi en innføring i noen
Foreleser: Eivind Coward, kontor 5. etg. Datablokken. coward@ii.uib.no Gruppeleder: Harald Barsnes Forelesninger: tirsdag og fredag 12 14 rom 2104 Øvinger: fredag 10 12 rom 2143 Gi en innføring i noen
Endret litt som ukeoppgave i INF1010 våren 2004
 Endret litt som ukeoppgave i INF1010 våren 2004!!!" # # $# ##!!%# # &##!'! Kontroller at oppgavesettet er komplett før du begynner å besvare det. Les gjerne gjennom hele oppgavesettet før du begynner med
Endret litt som ukeoppgave i INF1010 våren 2004!!!" # # $# ##!!%# # &##!'! Kontroller at oppgavesettet er komplett før du begynner å besvare det. Les gjerne gjennom hele oppgavesettet før du begynner med
INF 1040 Løsningsforslag til kapittel
 INF 040 Løsningsforslag til kapittel 8 Oppgave : Huffmankoding med kjente sannsynligheter Gitt en sekvens av symboler som er tilstrekkelig lang, og som inneholder de 6 symbolene A, B, C, D, E, F. Symbolene
INF 040 Løsningsforslag til kapittel 8 Oppgave : Huffmankoding med kjente sannsynligheter Gitt en sekvens av symboler som er tilstrekkelig lang, og som inneholder de 6 symbolene A, B, C, D, E, F. Symbolene
Sorteringsproblemet. Gitt en array A med n elementer som kan sammenlignes med hverandre:
 Sortering Sorteringsproblemet Gitt en array A med n elementer som kan sammenlignes med hverandre: Finn en ordning (eller permutasjon) av elementene i A slik at de står i stigende (evt. avtagende) rekkefølge
Sortering Sorteringsproblemet Gitt en array A med n elementer som kan sammenlignes med hverandre: Finn en ordning (eller permutasjon) av elementene i A slik at de står i stigende (evt. avtagende) rekkefølge
IN Algoritmer og datastrukturer
 IN00 - Algoritmer og datastrukturer HØSTEN 08 Institutt for informatikk, Universitetet i Oslo Forelesning 5: Grafer II Ingrid Chieh Yu (Ifi, UiO) IN00 8.09.08 / Dagens plan: Korteste vei en-til-alle vektet
IN00 - Algoritmer og datastrukturer HØSTEN 08 Institutt for informatikk, Universitetet i Oslo Forelesning 5: Grafer II Ingrid Chieh Yu (Ifi, UiO) IN00 8.09.08 / Dagens plan: Korteste vei en-til-alle vektet
UNIVERSITETET I BERGEN Det matematisk-naturvitenskapelige fakultet
 Skriftlig eksamen/written Examination Emne/Course: MOL204 Anvendt bioinformatikk I / Applied bioinformatics I Dato/Date: 16. desember/16 December Tillatte hjelpemidler (i samsvar med emnebeskrivelsen)/permitted
Skriftlig eksamen/written Examination Emne/Course: MOL204 Anvendt bioinformatikk I / Applied bioinformatics I Dato/Date: 16. desember/16 December Tillatte hjelpemidler (i samsvar med emnebeskrivelsen)/permitted
L12-Dataanalyse. Introduksjon. Nelson Aalen plott. Page 76 of Introduksjon til dataanalyse. Levetider og sensurerte tider
 Page 76 of 80 L12-Dataanalyse Introduksjon Introduksjon til dataanalyse Presentasjonen her fokuserer på dataanalyseteknikker med formål å estimere parametere (MTTF,, osv) i modeller vi benytter for vedlikeholdsoptimering
Page 76 of 80 L12-Dataanalyse Introduksjon Introduksjon til dataanalyse Presentasjonen her fokuserer på dataanalyseteknikker med formål å estimere parametere (MTTF,, osv) i modeller vi benytter for vedlikeholdsoptimering
Rekurrens. MAT1030 Diskret matematikk. Rekurrens. Rekurrens. Eksempel. Forelesning 16: Rekurrenslikninger. Dag Normann
 MAT1030 Diskret matematikk Forelesning 16: likninger Dag Normann Matematisk Institutt, Universitetet i Oslo INGEN PLENUMSREGNING 6/3 og 7/3 5. mars 008 MAT1030 Diskret matematikk 5. mars 008 Mandag ga
MAT1030 Diskret matematikk Forelesning 16: likninger Dag Normann Matematisk Institutt, Universitetet i Oslo INGEN PLENUMSREGNING 6/3 og 7/3 5. mars 008 MAT1030 Diskret matematikk 5. mars 008 Mandag ga
MOL204 Anvendt bioinformatikk I
 Universitetet i Bergen Molekylærbiologisk institutt Matematisk naturvitenskapelig embetseksamen MOL204 Anvendt bioinformatikk I bokmål / nynorsk / english Mandag 20. desember 2010, 4 timer, kl 9:00 13:00
Universitetet i Bergen Molekylærbiologisk institutt Matematisk naturvitenskapelig embetseksamen MOL204 Anvendt bioinformatikk I bokmål / nynorsk / english Mandag 20. desember 2010, 4 timer, kl 9:00 13:00
SIF8010 ALGORITMER OG DATASTRUKTURER
 SIF8010 ALGORITMER OG DATASTRUKTURER KONTINUASJONSEKSAMEN, 1999; LØSNINGSFORSLAG Oppgave 1 (12%) Anta at du skal lage et støtteprogram som umiddelbart skal varsle om at et ord blir skrevet feil under inntasting
SIF8010 ALGORITMER OG DATASTRUKTURER KONTINUASJONSEKSAMEN, 1999; LØSNINGSFORSLAG Oppgave 1 (12%) Anta at du skal lage et støtteprogram som umiddelbart skal varsle om at et ord blir skrevet feil under inntasting
Forelesning 30. Kompleksitetsteori. Dag Normann mai Informasjon. Oppsummering
 Forelesning 30 Kompleksitetsteori Dag Normann - 14. mai 2008 Informasjon Det er lagt ut program for orakeltjenestene i MAT1030 denne våren på semestersiden. Det blir ikke ordinære gruppetimer fra og med
Forelesning 30 Kompleksitetsteori Dag Normann - 14. mai 2008 Informasjon Det er lagt ut program for orakeltjenestene i MAT1030 denne våren på semestersiden. Det blir ikke ordinære gruppetimer fra og med
Dynamisk programmering Undervises av Stein Krogdahl
 Dynamisk programmering Undervises av Stein Krogdahl 5. september 2012 Dagens stoff er hentet fra kapittel 9 i læreboka, samt kapittel 20.5 (som vi «hoppet over» sist) Kapittel 9 er lagt ut på undervisningsplanen.
Dynamisk programmering Undervises av Stein Krogdahl 5. september 2012 Dagens stoff er hentet fra kapittel 9 i læreboka, samt kapittel 20.5 (som vi «hoppet over» sist) Kapittel 9 er lagt ut på undervisningsplanen.
INF Algoritmer og datastrukturer. Hva er INF2220? Algoritmer og datastrukturer
 Praktiske opplysninger INF2220 - Algoritmer og datastrukturer HØSTEN 2007 Institutt for informatikk, Universitetet i Oslo Tid og sted: Mandag kl. 12:15-14:00 Store auditorium, Informatikkbygningen Kursansvarlige
Praktiske opplysninger INF2220 - Algoritmer og datastrukturer HØSTEN 2007 Institutt for informatikk, Universitetet i Oslo Tid og sted: Mandag kl. 12:15-14:00 Store auditorium, Informatikkbygningen Kursansvarlige
Plenumsregning 9. Diverse ukeoppgaver. Roger Antonsen april Oppgaver fra forelesningene. Oppgave (fra forelesningen 10/3).
 Plenumsregning 9 Diverse ukeoppgaver Roger Antonsen - 10. april 2008 Oppgaver fra forelesningene Oppgave (fra forelesningen 10/3). a) Ved å bruke den rekursive definisjonen av PL, vis hvordan vi skritt
Plenumsregning 9 Diverse ukeoppgaver Roger Antonsen - 10. april 2008 Oppgaver fra forelesningene Oppgave (fra forelesningen 10/3). a) Ved å bruke den rekursive definisjonen av PL, vis hvordan vi skritt
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamensdag / Day of exam: 3. juni 2006/June 3 th 2006 Tid for eksamen / Exam hours: 900-200 Eksamen i / Exam in: MBV3070 Oppgavesettet
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamensdag / Day of exam: 3. juni 2006/June 3 th 2006 Tid for eksamen / Exam hours: 900-200 Eksamen i / Exam in: MBV3070 Oppgavesettet
Lineære likningssystemer og matriser
 Kapittel 3 Lineære likningssystemer og matriser I dette kapittelet skal vi sette sammen Kapittel 1 og 2. 3.1 Den utvidede matrisen til et likningssystem Vi starter med et lineært likningssystem med m likninger
Kapittel 3 Lineære likningssystemer og matriser I dette kapittelet skal vi sette sammen Kapittel 1 og 2. 3.1 Den utvidede matrisen til et likningssystem Vi starter med et lineært likningssystem med m likninger
MOL204 Anvendt bioinformatikk I
 Universitetet i Bergen Molekylærbiologisk institutt Matematisk-naturvitenskapelig Embetseksamen MOL204 Anvendt bioinformatikk I bokmål / nynorsk / english Mandag 15. desember 2008, 4 timer, kl 9:00-13:00
Universitetet i Bergen Molekylærbiologisk institutt Matematisk-naturvitenskapelig Embetseksamen MOL204 Anvendt bioinformatikk I bokmål / nynorsk / english Mandag 15. desember 2008, 4 timer, kl 9:00-13:00
Kodegenerering, del 2: Resten av Kap. 8 pluss tilleggsnotat (fra kap. 9 i ASU ) INF5110 V2007
 Kodegenerering, del 2: Resten av Kap. 8 pluss tilleggsnotat (fra kap. 9 i ASU ) INF5110 V2007 Stein Krogdahl, Ifi UiO NB: Innfører noen begreper som først og fremst har mening om man skal gå videre med
Kodegenerering, del 2: Resten av Kap. 8 pluss tilleggsnotat (fra kap. 9 i ASU ) INF5110 V2007 Stein Krogdahl, Ifi UiO NB: Innfører noen begreper som først og fremst har mening om man skal gå videre med
TDT4110 Informasjonsteknologi grunnkurs: Kapittel 7 Filer og unntak ( exceptions ) Professor Alf Inge Wang Stipendiat Lars Bungum
 1 TDT4110 Informasjonsteknologi grunnkurs: Kapittel 7 Filer og unntak ( exceptions ) Professor Alf Inge Wang Stipendiat Lars Bungum 2 Læringsmål Mål Introduksjon til filer (som inndata og utdata) Å bruke
1 TDT4110 Informasjonsteknologi grunnkurs: Kapittel 7 Filer og unntak ( exceptions ) Professor Alf Inge Wang Stipendiat Lars Bungum 2 Læringsmål Mål Introduksjon til filer (som inndata og utdata) Å bruke
Mer om Markov modeller
 Høyere ordens Markov modeller Mer om Markov modeller p h mnr = Pr( Y j+ 3 = ah Y j+ 2 = am, Y j+ 1 = an, Y j = a : r For en k-te ordens Markov modell som modellerer en DNA prosess vil det være 3*4 k mulige
Høyere ordens Markov modeller Mer om Markov modeller p h mnr = Pr( Y j+ 3 = ah Y j+ 2 = am, Y j+ 1 = an, Y j = a : r For en k-te ordens Markov modell som modellerer en DNA prosess vil det være 3*4 k mulige
TDT4102 Prosedyreog objektorientert programmering Vår 2016
 Norges teknisk naturvitenskapelige universitet Institutt for datateknikk og informasjonsvitenskap TDT4102 Prosedyreog objektorientert programmering Vår 2016 Øving 4 Frist: 2016-02-12 Mål for denne øvingen:
Norges teknisk naturvitenskapelige universitet Institutt for datateknikk og informasjonsvitenskap TDT4102 Prosedyreog objektorientert programmering Vår 2016 Øving 4 Frist: 2016-02-12 Mål for denne øvingen:
Repetisjon: operatorene ++ og -- Java 5. Nøtt. Oppgave 1 (fra forrige gang) 0 udefinert udefinert. Alternativ 1 Prefiks-operator
 Litt mer om løkker Arrayer le Christian Lingjærde Gruppen for bioinformatikk Institutt for informatikk Universitetet i slo Java Repetisjon: operatorene ++ og -- Instruksjon i = i + i = i - Alternativ Prefiks-operator
Litt mer om løkker Arrayer le Christian Lingjærde Gruppen for bioinformatikk Institutt for informatikk Universitetet i slo Java Repetisjon: operatorene ++ og -- Instruksjon i = i + i = i - Alternativ Prefiks-operator
INF Algoritmer og datastrukturer
 IN2220 - lgoritmer og datastrukturer HØSTN 2016 Institutt for informatikk, Universitetet i Oslo orelesning 7: rafer III Ingrid hieh Yu (Ifi, UiO) IN2220 05.10.2016 1 / 28 agens plan: evis for Prim ybde-først
IN2220 - lgoritmer og datastrukturer HØSTN 2016 Institutt for informatikk, Universitetet i Oslo orelesning 7: rafer III Ingrid hieh Yu (Ifi, UiO) IN2220 05.10.2016 1 / 28 agens plan: evis for Prim ybde-først
Drosjesentralen. I-120: Obligatorisk oppgave 2, 2000
 Drosjesentralen I-120: Obligatorisk oppgave 2, 2000 Frist Mandag 20. November 2000 kl.10:00, i skuff merket I120 på UA. Krav Se seksjon 4 for kravene til innlevering. Merk krav om generisk løsning for
Drosjesentralen I-120: Obligatorisk oppgave 2, 2000 Frist Mandag 20. November 2000 kl.10:00, i skuff merket I120 på UA. Krav Se seksjon 4 for kravene til innlevering. Merk krav om generisk løsning for
Eksamensoppgave i TDT4120 Algoritmer og datastrukturer
 Eksamensoppgave i TDT4120 Algoritmer og datastrukturer Faglig kontakt under eksamen Magnus Lie Hetland Tlf. 91851949 Eksamensdato 11. august 2014 Eksamenstid (fra til) 0900 1300 Hjelpemiddelkode D. Ingen
Eksamensoppgave i TDT4120 Algoritmer og datastrukturer Faglig kontakt under eksamen Magnus Lie Hetland Tlf. 91851949 Eksamensdato 11. august 2014 Eksamenstid (fra til) 0900 1300 Hjelpemiddelkode D. Ingen
Disjunkte mengder ADT
 Binære relasjoner A A = {(x, y) x, y A}: mengden av ordnede par over A. Disjunkte mengder ADT Weiss kap. 8.1 8.5 Løser ekvivalensproblemet Lett og rask implementasjon Vanskelig tidsforbrukanalyse Ark 1
Binære relasjoner A A = {(x, y) x, y A}: mengden av ordnede par over A. Disjunkte mengder ADT Weiss kap. 8.1 8.5 Løser ekvivalensproblemet Lett og rask implementasjon Vanskelig tidsforbrukanalyse Ark 1
Hva er en algoritme? INF HØSTEN 2006 INF1020. Kursansvarlige Ragnar Normann E-post: Dagens tema
 va er en algoritme? Vanlig sammenligning: Oppskrift. nput lgoritme NF1020 - ØSTEN 2006 Kursansvarlige Ragnar Normann E-post: ragnarn@ifi.uio.no Output Knuth : tillegg til å være et endelig sett med regler
va er en algoritme? Vanlig sammenligning: Oppskrift. nput lgoritme NF1020 - ØSTEN 2006 Kursansvarlige Ragnar Normann E-post: ragnarn@ifi.uio.no Output Knuth : tillegg til å være et endelig sett med regler
Definisjon: Et sortert tre
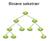 Binære søketrær Definisjon: Et sortert tre For alle nodene i et binært søketre gjelder: Alle verdiene i nodens venstre subtre er mindre enn verdien i noden Alle verdiene i nodens høyre subtre er større
Binære søketrær Definisjon: Et sortert tre For alle nodene i et binært søketre gjelder: Alle verdiene i nodens venstre subtre er mindre enn verdien i noden Alle verdiene i nodens høyre subtre er større
Oblig2 - obligatorisk oppgave nr. 2 (av 4) i INF1000
 Oblig2 - obligatorisk oppgave nr. 2 (av 4) i INF1000 Leveringsfrist Oppgaven må leveres senest fredag 30. september kl 16.00. Viktig: les slutten av oppgaven for detaljerte leveringskrav. Formål Formålet
Oblig2 - obligatorisk oppgave nr. 2 (av 4) i INF1000 Leveringsfrist Oppgaven må leveres senest fredag 30. september kl 16.00. Viktig: les slutten av oppgaven for detaljerte leveringskrav. Formål Formålet
4 Matriser TMA4110 høsten 2018
 Matriser TMA høsten 8 Nå har vi fått erfaring med å bruke matriser i et par forskjellige sammenhenger Vi har lært å løse et lineært likningssystem ved å sette opp totalmatrisen til systemet og gausseliminere
Matriser TMA høsten 8 Nå har vi fått erfaring med å bruke matriser i et par forskjellige sammenhenger Vi har lært å løse et lineært likningssystem ved å sette opp totalmatrisen til systemet og gausseliminere
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i Eksamensdag: 12. desember 2008 Tid for eksamen: 9.00 12.00 Oppgavesettet er på 7 sider. Vedlegg: Tillatte hjelpemidler: INF2220
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i Eksamensdag: 12. desember 2008 Tid for eksamen: 9.00 12.00 Oppgavesettet er på 7 sider. Vedlegg: Tillatte hjelpemidler: INF2220
Pattern matching algorithms. INF Algoritmer og datastrukturer. Lokalisering av Substrenger. Brute force
 Pattern matching algorithms INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo Algoritmer for lokalisering av substrenger Brute force Enkleste tenkelige
Pattern matching algorithms INF2220 - Algoritmer og datastrukturer HØSTEN 2009 Institutt for informatikk, Universitetet i Oslo Algoritmer for lokalisering av substrenger Brute force Enkleste tenkelige
Verdens statistikk-dag. Signifikanstester. Eksempel studentlån. http://unstats.un.org/unsd/wsd/
 Verdens statistikk-dag http://unstats.un.org/unsd/wsd/ Signifikanstester Ønsker å teste hypotese om populasjon Bruker data til å teste hypotese Typisk prosedyre Beregn sannsynlighet for utfall av observator
Verdens statistikk-dag http://unstats.un.org/unsd/wsd/ Signifikanstester Ønsker å teste hypotese om populasjon Bruker data til å teste hypotese Typisk prosedyre Beregn sannsynlighet for utfall av observator
LØSNINGSFORSLAG, EKSAMEN I ALGORITMER OG DATASTRUKTURER (IT1105)
 Norges teknisk naturvitenskapelige universitet Institutt for datateknikk og informasjonsvitenskap Side 1 av 8 Faglig kontakt under eksamen: Magnus Lie Hetland LØSNINGSFORSLAG, EKSAMEN I ALGORITMER OG DATASTRUKTURER
Norges teknisk naturvitenskapelige universitet Institutt for datateknikk og informasjonsvitenskap Side 1 av 8 Faglig kontakt under eksamen: Magnus Lie Hetland LØSNINGSFORSLAG, EKSAMEN I ALGORITMER OG DATASTRUKTURER
Ridge regresjon og lasso notat til STK2120
 Ridge regresjon og lasso notat til STK2120 Ørulf Borgan februar 2016 I dette notatet vil vi se litt nærmere på noen alternativer til minste kvadraters metode ved lineær regresjon. Metodene er særlig aktuelle
Ridge regresjon og lasso notat til STK2120 Ørulf Borgan februar 2016 I dette notatet vil vi se litt nærmere på noen alternativer til minste kvadraters metode ved lineær regresjon. Metodene er særlig aktuelle
Dagens stoff er hentet fra kapittel 9 i læreboka, samt kapittel 20.5 (som vi «hoppet over» sist)
 Dynamisk programmering Undervises av Stein Krogdahl 5. september 2012 Dagens stoff er hentet fra kapittel 9 i læreboka, samt kapittel 20.5 (som vi «hoppet over» sist) Kapittel 9 er lagt ut på undervisningsplanen.
Dynamisk programmering Undervises av Stein Krogdahl 5. september 2012 Dagens stoff er hentet fra kapittel 9 i læreboka, samt kapittel 20.5 (som vi «hoppet over» sist) Kapittel 9 er lagt ut på undervisningsplanen.
Hvor raskt klarer vi å sortere?
 Sortering Sorteringsproblemet Gitt en array med n elementer som kan sammenlignes med hverandre: Finn en ordning (eller permutasjon) av elementene slik at de står i stigende (evt. avtagende) rekkefølge
Sortering Sorteringsproblemet Gitt en array med n elementer som kan sammenlignes med hverandre: Finn en ordning (eller permutasjon) av elementene slik at de står i stigende (evt. avtagende) rekkefølge
MAT1030 Forelesning 28
 MAT1030 Forelesning 28 Kompleksitetsteori Roger Antonsen - 12. mai 2009 (Sist oppdatert: 2009-05-13 08:12) Forelesning 28: Kompleksitetsteori Introduksjon Da er vi klare (?) for siste kapittel, om kompleksitetsteori!
MAT1030 Forelesning 28 Kompleksitetsteori Roger Antonsen - 12. mai 2009 (Sist oppdatert: 2009-05-13 08:12) Forelesning 28: Kompleksitetsteori Introduksjon Da er vi klare (?) for siste kapittel, om kompleksitetsteori!
Dagens plan. INF3170 Logikk. Sekventkalkyle Gerhard Gentzen ( ) Innhold. Forelesning 12: Snitteliminasjon. Herman Ruge Jervell. 8.
 INF3170 Logikk Dagens plan Forelesning 12: Herman Ruge Jervell 1 Institutt for informatikk, Universitetet i Oslo 2 8. mai 2006 Institutt for informatikk (UiO) INF3170 Logikk 08.05.2006 2 / 27 Regler Innhold
INF3170 Logikk Dagens plan Forelesning 12: Herman Ruge Jervell 1 Institutt for informatikk, Universitetet i Oslo 2 8. mai 2006 Institutt for informatikk (UiO) INF3170 Logikk 08.05.2006 2 / 27 Regler Innhold
Anvendelser av grafer
 Grafer Anvendelser av grafer Passer for modeller/datastrukturer med usystematiske forbindelser Ikke-lineære og ikke-hierarkiske koblinger mellom dataobjektene Modellering av nettverk: Veisystemer/rutekart
Grafer Anvendelser av grafer Passer for modeller/datastrukturer med usystematiske forbindelser Ikke-lineære og ikke-hierarkiske koblinger mellom dataobjektene Modellering av nettverk: Veisystemer/rutekart
INF2220: Time 12 - Sortering
 INF0: Time 1 - Sortering Mathias Lohne mathialo Noen algoritmer Vi skal nå se på noen konkrete sorteringsalgoritmer. Gjennomgående i alle eksempler vil vi sortere tall etter tallverdi, men som diskutert
INF0: Time 1 - Sortering Mathias Lohne mathialo Noen algoritmer Vi skal nå se på noen konkrete sorteringsalgoritmer. Gjennomgående i alle eksempler vil vi sortere tall etter tallverdi, men som diskutert
IN1010 V18, Obligatorisk oppgave 5
 IN1010 V18, Obligatorisk oppgave 5 Innleveringsfrist: Tirsdag 17.04. kl 10:00 Versjon 1.3 (12.04.2018) Sist modifisert av Silje Merethe Dahl. Innledning I denne oppgaven skal du bruke rekursjon til å lage
IN1010 V18, Obligatorisk oppgave 5 Innleveringsfrist: Tirsdag 17.04. kl 10:00 Versjon 1.3 (12.04.2018) Sist modifisert av Silje Merethe Dahl. Innledning I denne oppgaven skal du bruke rekursjon til å lage
HØGSKOLEN I STAVANGER
 EKSAMEN I: MOT0 STATISTISKE METODER VARIGHET: TIMER DATO:. NOVEMBER 00 TILLATTE HJELPEMIDLER: KALKULATOR, TABELLER OG FORMLER I STATISTIKK (TAPIR FORLAG) OPPGAVESETTET BESTÅR AV OPPGAVER PÅ 7 SIDER HØGSKOLEN
EKSAMEN I: MOT0 STATISTISKE METODER VARIGHET: TIMER DATO:. NOVEMBER 00 TILLATTE HJELPEMIDLER: KALKULATOR, TABELLER OG FORMLER I STATISTIKK (TAPIR FORLAG) OPPGAVESETTET BESTÅR AV OPPGAVER PÅ 7 SIDER HØGSKOLEN
Dynamisk programmering
 Dynamisk programmering Metoden ble formalisert av Richard Bellmann (RAND Corporation) på 50-tallet. Programmering i betydningen planlegge, ta beslutninger. (Har ikke noe med kode eller å skrive kode å
Dynamisk programmering Metoden ble formalisert av Richard Bellmann (RAND Corporation) på 50-tallet. Programmering i betydningen planlegge, ta beslutninger. (Har ikke noe med kode eller å skrive kode å
MOL204 Anvendt bioinformatikk I
 Universitetet i Bergen Molekylærbiologisk institutt Matematisk-naturvitenskapelig embetseksamen MOL204 Anvendt bioinformatikk I bokmål / nynorsk / english Mandag 17. desember 2007, 4 timer, kl 9:00-13:00
Universitetet i Bergen Molekylærbiologisk institutt Matematisk-naturvitenskapelig embetseksamen MOL204 Anvendt bioinformatikk I bokmål / nynorsk / english Mandag 17. desember 2007, 4 timer, kl 9:00-13:00
To geometriske algoritmer, kap. 8.6
 INF 4130, 18. november 2010 To geometriske algoritmer, kap. 8.6 Computational Geometry Stein Krogdahl Hovedkapittelet t (kap. 8) dreier seg generelt om devide-and-conquer eller splitt og hersk : Splitt
INF 4130, 18. november 2010 To geometriske algoritmer, kap. 8.6 Computational Geometry Stein Krogdahl Hovedkapittelet t (kap. 8) dreier seg generelt om devide-and-conquer eller splitt og hersk : Splitt
UNIVERSITETET I OSLO. Det matematisk-naturvitenskapelige fakultet
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i: INF 4130: lgoritmer: Design og effektivitet Eksamensdag: 12. desember 2008 Tid for eksamen: Kl. 09:00 12:00 (3 timer) Oppgavesettet
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i: INF 4130: lgoritmer: Design og effektivitet Eksamensdag: 12. desember 2008 Tid for eksamen: Kl. 09:00 12:00 (3 timer) Oppgavesettet
GRAFER. Noen grafdefinisjoner. Korteste vei i en uvektet graf V 2 V 1 V 5 V 3 V 4 V 6
 IN Algoritmer og datastrukturer GRAER Dagens plan: Kort repetisjon om grafer Korteste, en-til-alle, for: uektede grafer (repetisjon) ektede rettede grafer uten negatie kanter (Dijkstra, kapittel 9..) ektede
IN Algoritmer og datastrukturer GRAER Dagens plan: Kort repetisjon om grafer Korteste, en-til-alle, for: uektede grafer (repetisjon) ektede rettede grafer uten negatie kanter (Dijkstra, kapittel 9..) ektede
Løse reelle problemer
 Løse reelle problemer Litt mer om løkker, metoder med returverdier og innlesing fra fil INF1000, uke4 Geir Kjetil Sandve Repetisjon fra forrige uke: while Syntaks: while (condition) do1; do2;... Eksempel:
Løse reelle problemer Litt mer om løkker, metoder med returverdier og innlesing fra fil INF1000, uke4 Geir Kjetil Sandve Repetisjon fra forrige uke: while Syntaks: while (condition) do1; do2;... Eksempel:
Oblig2 - obligatorisk oppgave nr. 2 (av 4) i INF1000 h2006
 Oblig2 - obligatorisk oppgave nr 2 (av 4) i INF1000 h2006 Leveringsfrist Oppgaven må leveres senest fredag 30 september kl 1600 Viktig: les slutten av oppgaven for detaljerte leveringskrav Formål Formålet
Oblig2 - obligatorisk oppgave nr 2 (av 4) i INF1000 h2006 Leveringsfrist Oppgaven må leveres senest fredag 30 september kl 1600 Viktig: les slutten av oppgaven for detaljerte leveringskrav Formål Formålet
