RelEx v. 1.0 (2004) Michael J. MacCoss and John D. Venable, The Scripps Research Institute, La Jolla, CA (2002)
|
|
|
- Ada Ask
- 5 år siden
- Visninger:
Transkript
1 RelEx v. 1.0 (2004) Michael J. MacCoss and John D. Venable, The Scripps Research Institute, La Jolla, CA (2002) P CE00015 locus:drs-1 Aspartyl-tRNA synthetase status:partially_confirm R U K.IISAPSEGGANVFEVSYFK.G _CelegD:\celegans\Celegans\rep1\chro\ R U K.YGCPPHAGGGIGLER.V _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.VTMLFLGLHNIR.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.IISAPSEGGANVFEVSYFK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.VTMLFLGLHNIR.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.IISAPSEGGANVFEVSYFK.G _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.VTMLFLGLHNIR.L _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00101 locus:rpn-3 Diphenol oxidase A2 status:confirmed SW:Q R U K.YLHYTPAADTEVQPMDTSNVK.T _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.YLHYTPAADTEVQPMDTSNVK.T _Celegd:\celegans\Celegans\rep2\chro\ P CE00103 Msi3p status:partially_confirmed SW:Q05036 protein_id:aaa R U R.IVEEVPAEAMEVDGDAK.T _CelegD:\celegans\Celegans\rep1\chro\ R U K.NSLEEYVYEMR.D _CelegD:\celegans\Celegans\rep1\chro\ R U R.VNPDGIFTIASATMYEPR.I _CelegD:\celegans\Celegans\rep1\chro\ R U R.IVNETTAIALAYGIYK.Q _CelegD:\celegans\Celegans\rep1\chro\ R U K.LPNDDIGVQVSYLGEPHTFTPEQVLAALLTK.L _CelegD:\celegans\Celegans\rep1\chro\ R U K.FHNLELQMQESDAR.E _CelegD:\celegans\Celegans\rep1\chro\ R U K.QMSANQTPIPLNIECFMEDK.D _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.KPAFDSFDQSIMR.V _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.VSDQYAEFITPAAADEFR.S _Celegd:\celegans\Celegans\rep2\chro\ S U K.NSLEEYVYEMR.D _Celegd:\celegans\Celegans\rep2\chro\ S U R.VNPDGIFTIASATMYEPR.I _Celegd:\celegans\Celegans\rep2\chro\ S U K.LPNDDIGVQVSYLGEPHTFTPEQVLAALLTK.L _Celegd:\celegans\Celegans\rep2\chro\ R U R.QEFEDLAAPIFNR.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.VSDCVLAVPSYFTDVQR.R _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.TDAPVVFTEEILQNK.N _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.IVNETTAIALAYGIYK.Q _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.FHNLELQMQESDAR.E _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.KPAFDSFDQSIMR.V _Celegd:\celegans\Celegans\rep2_N15\chro\ S U R.IVEEVPAEAMEVDGDAK.T _C_eled:\celegans\Celegans\rep3\chro\ S U R.LSWNSTGENGGENDVFSPR.D _C_eled:\celegans\Celegans\rep3\chro\ S U K.VSDQYAEFITPAAADEFR.S _C_eled:\celegans\Celegans\rep3\chro\ S U K.TDAPVVFTEEILQNK.N _C_eled:\celegans\Celegans\rep3\chro\ S U K.LVPVDLEVIESIPVSYDVQK.F _C_eled:\celegans\Celegans\rep3\chro\ S U R.VNPDGIFTIASATMYEPR.I _C_eled:\celegans\Celegans\rep3\chro\ S U K.LPNDDIGVQVSYLGEPHTFTPEQVLAALLTK.L _C_eled:\celegans\Celegans\rep3\chro\ R U R.QEFEDLAAPIFNR.I _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.VSDCVLAVPSYFTDVQR.R _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.FHNLELQMQESDAR.E _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.KPAFDSFDQSIMR.V _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00133 locus:far-1 O. volvulus 20Kd antigenic peptide status:confirm R U K.DSIAKEFPILTGFFKNEK.V _CelegD:\celegans\Celegans\rep1\chro\ R U K.THIEAYK.G _CelegD:\celegans\Celegans\rep1\chro\ R U K.IHAQYLAGEKPSLDTLK.T _CelegD:\celegans\Celegans\rep1\chro\ R U K.LHQIVK.D _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.KIHAQYLAGEKPSLDTLK.T _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.VQAMVGQYLN _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.DSIAKEFPILTGFFKNEK.V _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.THIEAYK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.IHAQYLAGEKPSLDTLK.T _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.KIHAQYLAGEKPSLDTLK.T _Celegd:\celegans\Celegans\rep2_N15\chro\ S U R.KIHAQYLAGEKPSLDTLK.T _C_eled:\celegans\Celegans\rep3\chro\ R U K.DSIAKEFPILTGFFKNEK.V _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.THIEAYK.G _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.IHAQYLAGEKPSLDTLK.T _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00134 locus:far-2 status:confirmed SW:P34383 protein_id:caa R U K.ATITAEFPILTGFFQNEK.I _CelegD:\celegans\Celegans\rep1\chro\ R U R.ELHQQYLAGDKPTLDSLK.E _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.ATITAEFPILTGFFQNEK.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.ELHQQYLAGDKPTLDSLK.E _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.ELHQQYLAGDKPTLDSLK.E _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00209 Membrane-associated atpase gamma chain status:confirmed R U R.IENTLTYIVTELDEMER.E _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE00213 locus:clu-1 status:confirmed SW:P34466 protein_id:caa R U R.AIPGFVDGCTLNVVDEPYTIR.D _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.VVVDAADFLLTQK.I _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00216 locus:ubc-7 Ubiquitin conjugating enzyme status:confirmed S S U K.FISEIWHPNIDK.E _Celegd:\celegans\Celegans\rep2\chro\ P CE00302 locus:gst-1 glutathione S-transferase status:confirmed SW:P R U K.LTYFDIHGLAEPIR.L _CelegD:\celegans\Celegans\rep1\chro\ R U R.VTYEQWADIKPK.M _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U R.VTYEQWADIKPK.M _Celegd:\celegans\Celegans\rep2\chro\ R U K.LTYFDIHGLAEPIR.L _Celegd:\celegans\Celegans\rep2_N15\chro\
2 S U K.LTYFDIHGLAEPIR.L _C_eled:\celegans\Celegans\rep3\chro\ P CE00319 status:partially_confirmed SW:P34560 protein_id:caa R U R.DYGLEAMDLDPSSFLAAK.W _CelegD:\celegans\Celegans\rep1\chro\ S U R.DYGLEAMDLDPSSFLAAK.W _Celegd:\celegans\Celegans\rep2\chro\ P CE00331 locus:rnr-1 Ribonucleoside-disphosphate reductase large cha S U K.LSYGLNMDFVDPVAVAIK.V _Celegd:\celegans\Celegans\rep2\chro\ S U K.IIDVNYYPVEEAR.N _C_eled:\celegans\Celegans\rep3\chro\ P CE00390 locus:lap-1 Leucine aminopeptidase status:partially_confirme R U K.LTNEDAVFLTDLSESVR.E _CelegD:\celegans\Celegans\rep1\chro\ S U R.WLHLDIAAPAEVGDR.G _Celegd:\celegans\Celegans\rep2\chro\ R U K.LTNEDAVFLTDLSESVR.E _Celegd:\celegans\Celegans\rep2_N15\chro\ S U K.LTNEDAVFLTDLSESVR.E _C_eled:\celegans\Celegans\rep3\chro\ R U R.DMGGAAGMLEAYSALVK.H _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.LIDTPANILTTDALVDEAVK.V _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.ASGDLVAPMLFAPDLFFGDLK.S _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00415 locus:mcm-6 Mcm2\/3 status:confirmed SW:P34647 protein_ S U K.NTIFINMQHLYK.F _Celegd:\celegans\Celegans\rep2\chro\ S U K.NTIFINMQHLYK.F _C_eled:\celegans\Celegans\rep3\chro\ P CE00480 Clathrin heavy chain status:partially_confirmed SW:P34574 p R U R.VGFQPDYLFQLR.Q _CelegD:\celegans\Celegans\rep1\chro\ R U K.LHVIEVGTPAAGNTPFQK.K _CelegD:\celegans\Celegans\rep1\chro\ S U R.SSIEAQDNFDNITLAQQLEK.H _Celegd:\celegans\Celegans\rep2\chro\ S U K.LHVIEVGTPAAGNTPFQK.K _Celegd:\celegans\Celegans\rep2\chro\ R U R.LLEMNLLAAPAVADAILANK.M _Celegd:\celegans\Celegans\rep2_N15\chro\ S U R.NGELAEELLSFFLDEK.L _C_eled:\celegans\Celegans\rep3\chro\ S U K.FDIDELVEEVEK.R _C_eled:\celegans\Celegans\rep3\chro\ S U R.NLQNLLILTAMR.A _C_eled:\celegans\Celegans\rep3\chro\ R U K.LHVIEVGTPAAGNTPFQK.K _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00503 locus:iff-1 Initiation factor 5A status:confirmed SW:P34563 p S N K.KLEDICPSTHNMDVPVVK.R _Celegd:\celegans\Celegans\rep2\chro\ S N K.KLEDICPSTHNMDVPVVK.R _C_eled:\celegans\Celegans\rep3\chro\ P CE00513 locus:cts-1 Citrate synthase status:confirmed SW:P34575 pr R U K.GGEEPLPEAIWWLLCTGDVPSEAQTAAITK.E _CelegD:\celegans\Celegans\rep1\chro\ R U R.MLDNFPDNLHPMAQFIAAIAALNNESK.F _CelegD:\celegans\Celegans\rep1\chro\ R U K.SGQVVPGYGHAVLR.K _CelegD:\celegans\Celegans\rep1\chro\ R U K.ASYWEYAYEDSMDLLAK.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.SGQVVPGYGHAVLR.K _Celegd:\celegans\Celegans\rep2\chro\ R U K.GGEEPLPEAIWWLLCTGDVPSEAQTAAITK.E _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.ASYWEYAYEDSMDLLAK.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.MLDNFPDNLHPMAQFIAAIAALNNESK.F _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.MLDNFPDNLHPMAQFIAAIAALNNESK.F _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.SGQVVPGYGHAVLR.K _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00548 Elongation factor 1 status:confirmed SW:P34460 protein_id:a R U R.WYANVASYTDAER.K _CelegD:\celegans\Celegans\rep1\chro\ R N K.ITGDFEDHVQSVDIVAFNK.I _CelegD:\celegans\Celegans\rep1\chro\ R U R.WYANVASYTDAER.K _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.SSVILDVKPWDDETDLGEMEK.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.ITGDFEDHVQSVDIVAFNK.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.WYANVASYTDAER.K _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.SSVILDVKPWDDETDLGEMEK.L _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.ITGDFEDHVQSVDIVAFNK.I _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00566 status:confirmed SW:P34748 protein_id:caa R U K.YDEPIRSIFYEEDVLR.A _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.YDEPIRSIFYEEDVLR.A _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE00638 UMP synthase status:partially_confirmed TR:Q18516 protein R U R.VSACSDLLNWTPGVNLDAK.S _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.LEINSELENLSSLPYVENVR.T _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.VSACSDLLNWTPGVNLDAK.S _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.VSACSDLLNWTPGVNLDAK.S _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.LEINSELENLSSLPYVENVR.T _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00657 Mer5 (mouse) status:confirmed SW:Q21824 protein_id:caa R U R.KDGGLGDMDIPLLADFNK.K _CelegD:\celegans\Celegans\rep1\chro\ R U K.IADSFGVLDKESGLSYR.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.HGEVCPADWHEDSPTIKPGVATSK.E _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.HGEVCPADWHEDSPTIKPGVATSK.E _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.IADSFGVLDKESGLSYR.G _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.HGEVCPADWHEDSPTIKPGVATSK.E _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00660 locus:cku-80 status:partially_confirmed TR:Q21829 protein_i S U R.TNQILDQIMAPKRR.V _Celegd:\celegans\Celegans\rep2\chro\ P CE00696 locus:arf-1 ARF status:confirmed SW:Q10943 protein_id:aa R N K.NISFTVWDVGGQDK.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.LGEIVTTIPTIGFNVETVEYK.N _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.NISFTVWDVGGQDK.I _Celegd:\celegans\Celegans\rep2_N15\chro\ S N K.NISFTVWDVGGQDK.I _C_eled:\celegans\Celegans\rep3\chro\ R N K.LGEIVTTIPTIGFNVETVEYK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00733 status:confirmed SW:P41994 protein_id:aaa R U R.YPNVPIEVLPLAAQPLLR.S _Celegd:\celegans\Celegans\rep2_N15\chro\
3 R U K.DIICVPTSFLTK.Q _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00748 locus:prs-1 Prolyl-tRNA synthetase region status:partially_co R U K.SLLDEIHTAMYNK.V _CelegD:\celegans\Celegans\rep1\chro\ R U K.THIADFAPEVAWVTR.A _CelegD:\celegans\Celegans\rep1\chro\ R U R.AFAWQNSWGLSTR.T _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.THIADFAPEVAWVTR.A _Celegd:\celegans\Celegans\rep2_N15\chro\ S U R.EFLWQEGHTAFANPADAEK.E _C_eled:\celegans\Celegans\rep3\chro\ S U K.THIADFAPEVAWVTR.A _C_eled:\celegans\Celegans\rep3\chro\ R U R.VAAVQVIVVPVGFK.T _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.SLLDEIHTAMYNK.V _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00804 status:partially_confirmed TR:Q21632 protein_id:aaa R U K.LFPDFQFTPFEQAIQESVQWFVDNYETAR.K _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE00827 status:confirmed SW:Q21993 protein_id:aak R U K.NCEQELNFFQESFNALK.G _CelegD:\celegans\Celegans\rep1\chro\ R U K.IATAGHTALIPLSESLYIR.A _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.NCEQELNFFQESFNALK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.IATAGHTALIPLSESLYIR.A _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.NCEQELNFFQESFNALK.G _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.IATAGHTALIPLSESLYIR.A _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00839 locus:cgh-1 ATP-dependent RNA helicase status:confirmed R U K.LLSQDFQGILDR.L _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE00850 locus:tbb-4 tubulin beta chain status:partially_confirmed SW: S U R.SLTVPELTQQMFDAK.N _Celegd:\celegans\Celegans\rep2\chro\ S U R.SLTVPELTQQMFDAK.N _C_eled:\celegans\Celegans\rep3\chro\ P CE hydroxyacyl-CoA dehydrogenase status:partially_confirmed R U K.INNVTIIGAGLMGSGIAQVSANAK.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.FIMDGWHAQYPEEVAFTPSPLLNSLVDSGK.N _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.INNVTIIGAGLMGSGIAQVSANAK.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.FIMDGWHAQYPEEVAFTPSPLLNSLVDSGK.N _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.LGAGYPMGPFELSDYVGLDTCK.F _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.FIMDGWHAQYPEEVAFTPSPLLNSLVDSGK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00868 NADPH-ferrihemoprotein reductase status:partially_confirme R N K.LCAIVVSSTGDGDAPDNCAR.F _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.CGVLDIADQQCELSINPK.T _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.AEADDQVGLELEVEPWIEK.F _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE00874 locus:rnr-2 Ribonuclease-diphosphate reductase M2 status:c S U K.NPFDFMENISIDGK.T _C_eled:\celegans\Celegans\rep3\chro\ S U R.NTLFNAVDEFEFIK.K _C_eled:\celegans\Celegans\rep3\chro\ P CE00897 locus:pdi-1 protein disulfide isomerase status:confirmed SW: R U K.SGPTVTTVESVEQLEELKGK.T _CelegD:\celegans\Celegans\rep1\chro\ R U K.LSAVTEFTHESAQEIVGGDLK.K _CelegD:\celegans\Celegans\rep1\chro\ R U K.VLVASNFNEIALDETK.T _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.FKPQEGEDFEAFTNSYLEGK.S _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.YAGSASESETASQDHEEL _Celegd:\celegans\Celegans\rep2\chro\ R U K.AQDLPEDWNALPVK.V _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.VLVASNFNEIALDETK.T _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.FKPQEGEDFEAFTNSYLEGK.S _Celegd:\celegans\Celegans\rep2_N15\chro\ S U K.AQDLPEDWNALPVK.V _C_eled:\celegans\Celegans\rep3\chro\ S U K.VLVASNFNEIALDETK.T _C_eled:\celegans\Celegans\rep3\chro\ S U K.SGPTVTTVESVEQLEELKGK.T _C_eled:\celegans\Celegans\rep3\chro\ S U K.FKPQEGEDFEAFTNSYLEGK.S _C_eled:\celegans\Celegans\rep3\chro\ P CE00913 locus:tbb-2 beta tubulin status:confirmed SW:P52275 protein R N R.AVLVDLEPGTMDSVR.S _CelegD:\celegans\Celegans\rep1\chro\ R N K.GHYTEGAELVDNVLDVIR.K _CelegD:\celegans\Celegans\rep1\chro\ R N K.IREEYPDRIMSSFSVVPSPK.V _CelegD:\celegans\Celegans\rep1\chro\ R N K.FWEVISDEHGIQPDGTFK.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.EAEGCDCLQGFQLTHSLGGGTGSGMGTLLISK.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.LTNPTYGDLNHLVSLTMSGVTTCLR.F _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N R.SGPFGQLFRPDNFVFGQSGAGNNWAK.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ S N R.IDVYYNEANNGK.Y _C_eled:\celegans\Celegans\rep3\chro\ S N R.EVDEQMLNVQNK.N _C_eled:\celegans\Celegans\rep3\chro\ S N R.AVLVDLEPGTMDSVR.S _C_eled:\celegans\Celegans\rep3\chro\ S N K.FWEVISDEHGIQPDGTFK.G _C_eled:\celegans\Celegans\rep3\chro\ S N K.GHYTEGAELVDNVLDVIR.K _C_eled:\celegans\Celegans\rep3\chro\ S N R.KEAEGCDCLQGFQLTHSLGGGTGSGMGTLLISK.I _C_eled:\celegans\Celegans\rep3\chro\ S N K.LTNPTYGDLNHLVSLTMSGVTTCLR.F _C_eled:\celegans\Celegans\rep3\chro\ S N R.SGPFGQLFRPDNFVFGQSGAGNNWAK.G _C_eled:\celegans\Celegans\rep3\chro\ S N R.LHFFMPGFAPLSAK.G _C_eled:\celegans\Celegans\rep3\chro\ R N R.IDVYYNEANNGK.Y _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.EAEGCDCLQGFQLTHSLGGGTGSGMGTLLISK.I _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.GHYTEGAELVDNVLDVIR.K _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01044 locus:rnp-4 RNA binding domain status:confirmed TR:Q R U K.EANEAIDQSNDTDLLGQNVK.V _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01192 status:confirmed TR:Q27317 protein_id:aaa status:c R N R.ITPVFGFVDPSGSK.D _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE01215 Hydratase-dehydrogenase-epimerase status:confirmed TR:Q R U R.VLHGEQYIEVYQPLSAEGK.L _Celegd:\celegans\Celegans\rep2_N15\chro\
4 R U R.FSSPVLPGQTLVTETWK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.TPILHGLCSLGFATR.H _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01217 Isovaleryl-CoA dehydrogenase status:confirmed TR:Q S U R.VTGGNTGSNFGQVVDASLQDSAK.V _C_eled:\celegans\Celegans\rep3\chro\ P CE01225 Fructose-biphosphate aldolase status:confirmed SW:P R N K.DELHQIALK.I _CelegD:\celegans\Celegans\rep1\chro\ R N K.RLDAINLENNETNR.Q _CelegD:\celegans\Celegans\rep1\chro\ R N K.DSLTQAQKDELHQIALK.I _CelegD:\celegans\Celegans\rep1\chro\ R U K.ASHEAIGLATVTALR.R _CelegD:\celegans\Celegans\rep1\chro\ R N R.QLLFTTPNLNQHISGVILYEETFHQSTDK.G _CelegD:\celegans\Celegans\rep1\chro\ R U K.YAGEDAAGAAAESLFVAK.H _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.YAGEDAAGAAAESLFVAK.H _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.DELHQIALK.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R N R.YASICQANGLVPIVEPEVLCDGEHDLAR.A _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.RLDAINLENNETNR.Q _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.ASHEAIGLATVTALR.R _Celegd:\celegans\Celegans\rep2_N15\chro\ R N R.CVLNIGTHTPSHLGMLENANVLAR.Y _Celegd:\celegans\Celegans\rep2_N15\chro\ S U K.YAGEDAAGAAAESLFVAK.H _C_eled:\celegans\Celegans\rep3\chro\ R U K.ASHEAIGLATVTALR.R _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01231 status:partially_confirmed TR:Q19086 protein_id:aac R U K.NQNVIAHGADFYAYPR.V _CelegD:\celegans\Celegans\rep1\chro\ P CE01232 locus:alh-9 Aldehyde dehydrogenase status:confirmed SW:P R U R.IGCPLDSNTIIGPLHNQQAVGK.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.DGNFVLPTIVTGLK.H _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01234 locus:cct-6 status:confirmed SW:P46550 protein_id:aac R U K.LGAEAFAQALLVIPK.T _CelegD:\celegans\Celegans\rep1\chro\ S U K.LGAEAFAQALLVIPK.T _Celegd:\celegans\Celegans\rep2\chro\ P CE01235 status:partially_confirmed SW:Q19087 protein_id:aac R U K.AAIVGMEDEFISGAR.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.SMLISADQAHATHPNYSAK.H _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.AAIVGMEDEFISGAR.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.SMLISADQAHATHPNYSAK.H _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01270 locus:eft-3 Elongation factor 1-alpha status:confirmed SW:P R N R.LPLQDVYK.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N R.FTEITNEVSGFIK.K _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.IGGIGTVPVGR.V _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.YYITIIDAPGHR.D _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.STTTGHLIYK.C _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.FLKSGDAGIVELIPTKPLCVESFTDYAPLGR.F _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.TLLEALDSIIPPQRPTDRPLR.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.QLIVACNK.M _Celegd:\celegans\Celegans\rep2_N15\chro\ R N R.FTEITNEVSGFIK.K _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.AVPFVPISGFNGDNMLEVSSNMPWFK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.YYITIIDAPGHR.D _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.STTTGHLIYK.C _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.FLKSGDAGIVELIPTKPLCVESFTDYAPLGR.F _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.TLLEALDSIIPPQRPTDRPLR.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.AVPFVPISGFNGDNMLEVSSNMPWFK.G _C_eled:\celegans\Celegans\rep3_N15\chro\ R N R.FTEITNEVSGFIK.K _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.YYITIIDAPGHR.D _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.STTTGHLIYK.C _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.FLKSGDAGIVELIPTKPLCVESFTDYAPLGR.F _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.TLLEALDSIIPPQRPTDRPLR.L _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01296 status:confirmed TR:Q20310 protein_id:aaa R U K.ATEEPEQAVTISDK.A _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01301 locus:cyp-6 status:confirmed SW:P52014 protein_id:aaa R U R.VIENFMIQGGDFTR.G _CelegD:\celegans\Celegans\rep1\chro\ P CE01341 locus:inf-1 status:confirmed SW:P27639 protein_id:aaa R U K.MFVLDEADEMLSR.G _CelegD:\celegans\Celegans\rep1\chro\ R U K.MTENQFTVSCLHGDMDQAER.D _CelegD:\celegans\Celegans\rep1\chro\ R U R.IDHEDPHVQALVMAPTR.E _CelegD:\celegans\Celegans\rep1\chro\ S N R.GIYGFGFEKPSAIQK.R _Celegd:\celegans\Celegans\rep2\chro\ R U R.IDHEDPHVQALVMAPTR.E _Celegd:\celegans\Celegans\rep2_N15\chro\ S N R.GIYGFGFEKPSAIQK.R _C_eled:\celegans\Celegans\rep3\chro\ R U K.MFVLDEADEMLSR.G _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.MTENQFTVSCLHGDMDQAER.D _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.IDHEDPHVQALVMAPTR.E _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01436 status:confirmed SW:Q23280 protein_id:aag R U K.TDKYEATLEFFDEIDPASVK.H _CelegD:\celegans\Celegans\rep1\chro\ R U K.TDKYEATLEFFDEIDPASVK.H _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.TDKYEATLEFFDEIDPASVK.H _Celegd:\celegans\Celegans\rep2\chro\ S U K.LHFQGSSK.T _Celegd:\celegans\Celegans\rep2\chro\ S U K.TDKYEATLEFFDEIDPASVK.H _C_eled:\celegans\Celegans\rep3\chro\ S U K.LHFQGSSK.T _C_eled:\celegans\Celegans\rep3\chro\ P CE oxoacid CoA-transferase status:confirmed SW:Q09450 pro R U K.LLVGGFGLCGIPENLIQAITK.T _CelegD:\celegans\Celegans\rep1\chro\ R U R.QYLSGELELEFTPQGTLAER.I _CelegD:\celegans\Celegans\rep1_N15\chro1\
5 R U K.ILEHCELPLTGK.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.QYLSGELELEFTPQGTLAER.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.ILEHCELPLTGK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.IITDMAVFDVDTK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.ILEHCELPLTGK.G _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE01531 locus:mlc-4 myosin regulatory light chain 2 status:confirmed R U R.YSEEQVDELFR.D _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE01640 status:confirmed SW:Q09347 protein_id:caa S U R.ACFNFITYQVEK.R _Celegd:\celegans\Celegans\rep2\chro\ P CE01690 status:confirmed SW:Q09359 protein_id:caa R N R.SGEQPLYLEWFK.K _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N R.SGEQPLYLEWFK.K _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE01707 acetate utilisation protein status:confirmed TR:Q09657 prote R U R.MSHLILSGNCPQFDPK.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.MSHLILSGNCPQFDPK.Y _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE01733 locus:pas-7 proteasome component (A-type) status:confirme R U K.LKIEELDVNQLVK.E _CelegD:\celegans\Celegans\rep1\chro\ R U R.LFLVEPSGLNYEYK.A _CelegD:\celegans\Celegans\rep1_N15\chro1\ P CE01822 tryptophan 2,3-dioxygenase status:confirmed SW:Q09474 pr R N K.SLLTLIESWLER.T _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.SLLTLIESWLER.T _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE01917 Tripeptidyl-peptidase II status:partially_confirmed SW:Q S U K.GANYTEIVGIDTADPSLGPIFR.I _Celegd:\celegans\Celegans\rep2\chro\ P CE02065 locus:snr-3 small nuclear ribonucleoprotein Sm D1 (snrnp c R U R.YIILPDPLALDTLLIDDEPR.K _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02114 transitional endoplasmic reticulum ATPase homolog 1 (P97 p R U R.IVSQLLTLMDGVK.G _CelegD:\celegans\Celegans\rep1\chro\ S U K.DLDLTFLAK.N _Celegd:\celegans\Celegans\rep2\chro\ S N R.EAVVETPNTTWSDIGGLQNVK.R _Celegd:\celegans\Celegans\rep2\chro\ S N R.EAVVETPNTTWSDIGGLQNVK.R _Celegd:\celegans\Celegans\rep2\chro\ S U K.ESVAIIVSDESCPNEK.V _Celegd:\celegans\Celegans\rep2\chro\ S N R.AAAPCVLFFDELDSIAK.A _Celegd:\celegans\Celegans\rep2\chro\ S U K.NQPAILFIDEIDAIAPK.R _Celegd:\celegans\Celegans\rep2\chro\ S N R.AAAPCVLFFDELDSIAKAR.G _Celegd:\celegans\Celegans\rep2\chro\ S N K.SSPSALREAVVETPNTTWSDIGGLQNVKR.E _Celegd:\celegans\Celegans\rep2\chro\ R U R.IVSQLLTLMDGVK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ S U R.GEELMEDDAVDPVPEITR.A _C_eled:\celegans\Celegans\rep3\chro\ S N R.ELQELVQYPVEHPEK.Y _C_eled:\celegans\Celegans\rep3\chro\ S U R.LGDVVSITPAPNLSYGTR.I _C_eled:\celegans\Celegans\rep3\chro\ S N R.VINQVLTEMDGMNAK.K _C_eled:\celegans\Celegans\rep3\chro\ S N R.AAAPCVLFFDELDSIAK.A _C_eled:\celegans\Celegans\rep3\chro\ S U K.NQPAILFIDEIDAIAPK.R _C_eled:\celegans\Celegans\rep3\chro\ R N R.EAVVETPNTTWSDIGGLQNVK.R _C_eled:\celegans\Celegans\rep3_N15\chro\ R N R.LDQLIYIPLPDEASR.H _C_eled:\celegans\Celegans\rep3_N15\chro\ R N R.KYEMFAQTLQQSR.G _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02138 status:confirmed TR:Q17949 protein_id:caa R U R.HEVAYVLGQLQSPVATQELK.D _CelegD:\celegans\Celegans\rep1\chro\ S U K.FSDAEIDSFGEALNDTK.K _C_eled:\celegans\Celegans\rep3\chro\ S U R.HEVAYVLGQLQSPVATQELK.D _C_eled:\celegans\Celegans\rep3\chro\ R U R.HEAGEALGAIADPSVK.D _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02181 locus:ldh-1 L-lactate dehydrogenase status:confirmed SW:Q R U K.GFHGINDDVYLSLPVVLGSAGLTHVVK.Q _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.GFHGINDDVYLSLPVVLGSAGLTHVVK.Q _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE02183 locus:alh-8 methylmalonate-semialdehyde dehydrogenase st R U K.NLKVNAGWKPDTDIGPLISK.Q _CelegD:\celegans\Celegans\rep1\chro\ R U R.CMALTTAVLVGEAR.A _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.VNAGWKPDTDIGPLISK.Q _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.TVTQYWNESLTELKPQMSFPQLK _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.NHGVIMADANK.E _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.NAFNTWKNTSPLTR.Q _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.KFTNEVDVGQIGINVPIPVPLPMFSFTGSR.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.CMALTTAVLVGEAR.A _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.VNAGWKPDTDIGPLISK.Q _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.TVTQYWNESLTELKPQMSFPQLK _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.NAFNTWKNTSPLTR.Q _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.NLKVNAGWKPDTDIGPLISK.Q _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.KFTNEVDVGQIGINVPIPVPLPMFSFTGSR.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.VNAGWKPDTDIGPLISK.Q _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.TVTQYWNESLTELKPQMSFPQLK _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.NAFNTWKNTSPLTR.Q _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.NLKVNAGWKPDTDIGPLISK.Q _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.KFTNEVDVGQIGINVPIPVPLPMFSFTGSR.G _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02193 locus:pab-2 poly-adenylate binding protein status:confirmed R N R.LSLGYAYVNFQQPADAER.A _CelegD:\celegans\Celegans\rep1\chro\ P CE02218 adenylate kinase status:confirmed SW:Q20140 protein_id:ca R U R.GAQLTAIMESGALVPLEVVLDLVK.E _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.GAQLTAIMESGALVPLEVVLDLVK.E _C_eled:\celegans\Celegans\rep3_N15\chro\
6 P CE02249 locus:iff-2 initiation factor 5A status:confirmed SW:Q20751 p R N K.KLEDICPSTHNMDVPVVK.R _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.KLEDICPSTHNMDVPVVK.R _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.KLEDICPSTHNMDVPVVK.R _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02262 locus:cct-4 T-complex protein status:confirmed SW:P47208 p R U K.AQDIEAGDGTTTVVVMAGALLDAAQNLLSK.G _CelegD:\celegans\Celegans\rep1\chro\ R U K.AQDIEAGDGTTTVVVMAGALLDAAQNLLSK.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.VTGVQNPGHAVSILLR.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.AQDIEAGDGTTTVVVMAGALLDAAQNLLSK.G _Celegd:\celegans\Celegans\rep2\chro\ R U R.NLAQTQHGATQYCWR.A _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.VTGVQNPGHAVSILLR.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.VVSQHSWLLAPMAVNAVK.K _Celegd:\celegans\Celegans\rep2_N15\chro\ S U K.ILDEMSSPVDLSNDALLNK.M _C_eled:\celegans\Celegans\rep3\chro\ S U K.TDMENQVIITDYAQMDR.A _C_eled:\celegans\Celegans\rep3\chro\ S U K.AQDIEAGDGTTTVVVMAGALLDAAQNLLSK.G _C_eled:\celegans\Celegans\rep3\chro\ S U K.VVSQHSWLLAPMAVNAVK.K _C_eled:\celegans\Celegans\rep3\chro\ R U K.VTGVQNPGHAVSILLR.G _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02290 thioredoxin status:confirmed TR:Q21763 protein_id:caa R U R.EYFLEHMGAWTAIPFGTPR.I _CelegD:\celegans\Celegans\rep1\chro\ S U R.EYFLEHMGAWTAIPFGTPR.I _Celegd:\celegans\Celegans\rep2\chro\ S U K.LINQDSEELDAGEHLK.G _C_eled:\celegans\Celegans\rep3\chro\ S U R.EYFLEHMGAWTAIPFGTPR.I _C_eled:\celegans\Celegans\rep3\chro\ P CE02319 locus:cct-1 T-complex protein status:confirmed SW:P41988 p S U K.IIGPDADFFGELVVDAAEAVR.V _Celegd:\celegans\Celegans\rep2\chro\ S U K.MLVDDVGDVIVTNDGATILK.Q _C_eled:\celegans\Celegans\rep3\chro\ S U K.IIGPDADFFGELVVDAAEAVR.V _C_eled:\celegans\Celegans\rep3\chro\ P CE02329 locus:rpn-9 status:confirmed TR:Q22253 protein_id:caa S U R.TQIDSTQHEVDSLVGVTEVHAPFYR.V _C_eled:\celegans\Celegans\rep3\chro\ P CE02343 locus:gpd-1 glyceraldehyde 3-phosphate dehydrogenase 1 st R N K.VIISAPSADAPMYVVGVNHEK.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.KVIISAPSADAPMYVVGVNHEK.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ S N K.DPAAIAWGSVK.A _Celegd:\celegans\Celegans\rep2\chro\ S N K.ADFVVESTGVFTTK.E _Celegd:\celegans\Celegans\rep2\chro\ S N K.LVSWYDNEYGYSNR.V _Celegd:\celegans\Celegans\rep2\chro\ S N K.GTVTYDGDFLIVQK.D _Celegd:\celegans\Celegans\rep2\chro\ S N R.VPTPDVSVVDLTVR.L _Celegd:\celegans\Celegans\rep2\chro\ S N K.YDSTHGQFK.G _Celegd:\celegans\Celegans\rep2\chro\ S N K.VIISAPSADAPMYVVGVNHEK.Y _Celegd:\celegans\Celegans\rep2\chro\ S N K.KVIISAPSADAPMYVVGVNHEK.Y _Celegd:\celegans\Celegans\rep2\chro\ S N K.DTVQVVAVNDPFITIDYMVYLFK.Y _C_eled:\celegans\Celegans\rep3\chro\ S N K.ADFVVESTGVFTTK.E _C_eled:\celegans\Celegans\rep3\chro\ S N K.GTVTYDGDFLIVQK.D _C_eled:\celegans\Celegans\rep3\chro\ S N K.LVSWYDNEYGYSNR.V _C_eled:\celegans\Celegans\rep3\chro\ S N R.VPTPDVSVVDLTVR.L _C_eled:\celegans\Celegans\rep3\chro\ S N K.VIISAPSADAPMYVVGVNHEK.Y _C_eled:\celegans\Celegans\rep3\chro\ S N K.KVIISAPSADAPMYVVGVNHEK.Y _C_eled:\celegans\Celegans\rep3\chro\ P CE02347 locus:hpd-1 4-hydroxyphenylpyruvate dioxygenase status:co R U R.GCEFLSIPSSYYDNLK.E _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.AAGATIVHDITEESDADGSIR.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.GCEFLSIPSSYYDNLK.E _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.AAGATIVHDITEESDADGSIR.Y _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.FWSVDDSMIHTEYSALR.S _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.AAGATIVHDITEESDADGSIR.Y _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.FWSVDDSMIHTEYSALR.S _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02372 glutamate synthase status:partially_confirmed TR:Q22275 pr R U R.LGGATFEILAAEALK.R _CelegD:\celegans\Celegans\rep1\chro\ P CE02404 H+-transporting ATPase status:confirmed SW:Q23680 prote R U K.QTTVQEIEEAFNGFCAR.D _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.DDIAIILINQHIAEMIR.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.DDIAIILINQHIAEMIR.Y _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE k antigen (O. volvulus) status:confirmed TR:Q17698 prote R U K.GQQGQAEAAHQAAVSNFSPAAK.K _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.ADADLTAISNDSSLSVQAK.G _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.KADADLTAISNDSSLSVQAK.G _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02477 Aspartate aminotransferase status:confirmed TR:Q17994 pro R N K.LSAQLALGENSDVIK.N _CelegD:\celegans\Celegans\rep1\chro\ R U R.NWEHITNQIGMFCFTGINPQQVEK.L _CelegD:\celegans\Celegans\rep1\chro\ R U R.VGAFSVVTSDADEAAR.V _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N R.IFTTQSISGTGALR.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.SWWSHVEMGPPDAILGVTEAFK.A _CelegD:\celegans\Celegans\rep1_N15\chro1\ S N K.EYAGIVGLPEFTK.L _Celegd:\celegans\Celegans\rep2\chro\ S N K.LSAQLALGENSDVIK.N _Celegd:\celegans\Celegans\rep2\chro\ R U R.VGAFSVVTSDADEAAR.V _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.SWWSHVEMGPPDAILGVTEAFK.A _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.NWEHITNQIGMFCFTGINPQQVEK.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.VIYQPTPTWGNHVPIFK.F _Celegd:\celegans\Celegans\rep2_N15\chro\ S U R.VGAFSVVTSDADEAAR.V _C_eled:\celegans\Celegans\rep3\chro\
7 S N K.EYAGIVGLPEFTK.L _C_eled:\celegans\Celegans\rep3\chro\ R U R.YFVEQGHNIVLSQSFAK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.SWWSHVEMGPPDAILGVTEAFK.A _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.NWEHITNQIGMFCFTGINPQQVEK.L _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02496 status:partially_confirmed SW:Q18164 protein_id:aak R U K.WVFAGGDVAGVAETTVESVNDGK.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.FENPFGLASAPPTTSGPMCR.R _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.FENPFGLASAPPTTSGPMCR.R _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02568 status:confirmed TR:Q18886 protein_id:aaa S U R.EALENASTNAEVLNVMGTASK.A _Celegd:\celegans\Celegans\rep2\chro\ P CE02603 locus:msp-74 status:predicted TR:Q95PJ7 protein_id:aaa R N R.ITVEWTNTPDGAAK.Q _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N R.REWFQGDGMVR.R _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.EAVLLAVSCDAFAFGQEDTNNDR.I _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE02610 Deoxyribose-phosphate aldolase status:partially_confirmed S R U R.ASWASILAGSDFIK.T _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.LNSNITSVAGGFPSGQYHLK.S _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.TVDEALSYVALVK.D _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.ASWASILAGSDFIK.T _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.DILGDEWLNPHLFR.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.LNSNITSVAGGFPSGQYHLK.S _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.LNSNITSVAGGFPSGQYHLK.S _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.TVDEALSYVALVK.D _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.ASWASILAGSDFIK.T _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.LNSNITSVAGGFPSGQYHLK.S _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02632 locus:rpn-5 status:partially_confirmed TR:Q19324 protein_id R U R.LAELLDFPVDEMESFVCNLIVTGQITGAK.L _CelegD:\celegans\Celegans\rep1\chro\ P CE02699 locus:msp-3 Major sperm protein status:confirmed SW:Q R N K.IVFNAPYDDKHTYHIK.V _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02733 Serine carboxypeptidase status:confirmed SW:P52717 prote S U R.APQMYYAVQQFLNNHPL _Celegd:\celegans\Celegans\rep2\chro\ P CE02751 killer toxin-resistance protein (S. cerevisiae) status:partially_c S U K.FSDASFSDVFGVNSDYDK.G _C_eled:\celegans\Celegans\rep3\chro\ P CE02811 locus:msp-63 Major sperm protein status:partially_confirmed R N R.REWFQGDGMVR.R _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02816 status:partially_confirmed TR:Q21392 protein_id:aac R U K.NAVINVPEHTGTPLR.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.ADGSPDAETLQASQFPASGAK.L _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.NAVINVPEHTGTPLR.I _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02846 Glycine dehydrogenase status:partially_confirmed TR:Q R U R.FGVPLGYGGPHAGFMAVAK.H _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE02985 locus:cct-5 TCP-1 like chaperonin status:confirmed SW:P S U K.ALETLDSISDKFPVENR.E _C_eled:\celegans\Celegans\rep3\chro\ P CE03007 histone binding protein status:partially_confirmed TR:Q R U K.IEALEAEQSGISAIEEWNLK.L _CelegD:\celegans\Celegans\rep1\chro\ P CE03025 locus:hel-1 translation initiation factor eif-4a status:partially_ R N R.LANDCPHIVVGTPGR.M _CelegD:\celegans\Celegans\rep1\chro\ R U R.VNIVFNYDMPEDSDSYLHR.V _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U R.VNIVFNYDMPEDSDSYLHR.V _Celegd:\celegans\Celegans\rep2\chro\ S N K.TAVFVITTLQQLEPVDGEVSVVCMCHTR.E _Celegd:\celegans\Celegans\rep2\chro\ S U R.CEALHQLLTEQNFPSIAIHR.Q _Celegd:\celegans\Celegans\rep2\chro\ S U R.VNIVFNYDMPEDSDSYLHR.V _C_eled:\celegans\Celegans\rep3\chro\ S U R.CEALHQLLTEQNFPSIAIHR.Q _C_eled:\celegans\Celegans\rep3\chro\ P CE03076 acetyl-coenzyme A synthetase status:confirmed TR:Q R U K.VAEAAVVAAPHDIK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.IAEGSESGIGDTTTLVDESVIK.Q _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.TYLAPFNGYYFTGDGAR.R _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03099 locus:gst-42 glutathione S-transferase status:partially_confirm R U R.FNLDLSPYPTVNR.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.AISLLVASGIQPLHNLK.V _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.AISLLVASGIQPLHNLK.V _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE03105 locus:pyr-1 glutamine-dependent carbamoyl-phosphate synth R U R.NSVTGTTTACFEPSLDYCVVK.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.AVHIAHVATADEINLVK.E _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03244 phosphoacetylglucosamine mutase status:partially_confirmed S U R.VINETVGDAFADLLAVEAVLR.H _Celegd:\celegans\Celegans\rep2\chro\ S U R.VYAEADTVENTLQLGK.S _Celegd:\celegans\Celegans\rep2\chro\ S U K.FAEPTHDGYYSAIADSFK.K _C_eled:\celegans\Celegans\rep3\chro\ S U R.VINETVGDAFADLLAVEAVLR.H _C_eled:\celegans\Celegans\rep3\chro\ P CE03352 histidine ammonia-lyase status:partially_confirmed SW:Q R U K.MWSPTTGWQPADVVLK.K _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.ALDFLAIAVAELAQMSER.R _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03368 locus:rpn-7 status:confirmed SW:Q20585 protein_id:caa S U K.IDEITAEVEDAEK.N _C_eled:\celegans\Celegans\rep3\chro\ S U K.EFDMAPFYELVCADFK.I _C_eled:\celegans\Celegans\rep3\chro\ P CE03436 isocitrate dehydrogenase status:confirmed TR:Q21032 prote R U K.DIFAEIYPEYEAEFK.A _CelegD:\celegans\Celegans\rep1\chro\
8 R U K.FVSADGTQTIQETVFDFK.G _CelegD:\celegans\Celegans\rep1\chro\ R U R.TDYLNTFEFLDK.L _CelegD:\celegans\Celegans\rep1\chro\ R U K.GQETSTNPIASIFAWSR.G _CelegD:\celegans\Celegans\rep1\chro\ R U K.IQGGDIVEMQGDEMTR.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.GPGVSLSMYNTDDSIR.D _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.DFAHASFK.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.NILGGTVFREPIIVK.N _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.KFPLYLSTK.N _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.LVNTWSKPIIIGR.H _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.FVSADGTQTIQETVFDFK.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.GQETSTNPIASIFAWSR.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R N R.NILGGTVFR.E _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.DFAHASFK.Y _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.NILGGTVFREPIIVK.N _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.KFPLYLSTK.N _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.LVNTWSKPIIIGR.H _Celegd:\celegans\Celegans\rep2_N15\chro\ S U K.FVSADGTQTIQETVFDFK.G _C_eled:\celegans\Celegans\rep3\chro\ S U R.TDYLNTFEFLDK.L _C_eled:\celegans\Celegans\rep3\chro\ R U R.DATDDQVTIDAANATLK.Y _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.IQGGDIVEMQGDEMTR.I _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.NILGGTVFREPIIVK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ R U R.LVNTWSKPIIIGR.H _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03457 locus:eca-39 twt1 like protein status:confirmed SW:P54688 p R U K.VTDEYEELHIPTMSSK.F _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.AALPDFDSEEMINVLTELLR.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.FGHTYADYMMTCDWDAER.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.AALPDFDSEEMINVLTELLR.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.AALPDFDSEEMINVLTELLR.L _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03482 locus:let-70 ubiquitin-conjugating enzyme E2-17 KD status:c R U R.IYHPNINSNGSICLDILR.S _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE03567 locus:asp-4 aspartyl protease status:partially_confirmed TR:Q S U K.FDGILGMAYPEIAVLGVQPVFNTLFEQK.K _C_eled:\celegans\Celegans\rep3\chro\ P CE03659 heat shock protein status:partially_confirmed TR:Q22515 pro R U K.IGLVKPGVPMDIVLNK.E _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.TDNIDLNEEEVTAAK.K _C_eled:\celegans\Celegans\rep3\chro\ P CE03684 enolase status:confirmed SW:Q27527 protein_id:caa R N R.GNPTVEVDLFTEK.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N R.IEEELGADAVYAGHNFR.N _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.WLSGEQLTELYQSFIK.E _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.IAPALIAK.G _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.GNLGANAILGVSLAVAK.A _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.LAMQEFMILPVGASSFAEAMR.M _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.FHGATSIQLVGDDLTVTNPK.R _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.VVLPVPAFNVINGGSHAGNK.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.LAMQEFMILPVGASSFAEAMR.M _Celegd:\celegans\Celegans\rep2\chro\ R U K.VVLPVPAFNVINGGSHAGNK.L _Celegd:\celegans\Celegans\rep2_N15\chro\ S N K.WLSGEQLTELYQSFIK.E _C_eled:\celegans\Celegans\rep3\chro\ R U R.YGLDATAVGDEGGFAPNIQDNK.E _C_eled:\celegans\Celegans\rep3_N15\chro\ R N R.SGETEDTFIADLVVGLATGQIK.T _C_eled:\celegans\Celegans\rep3_N15\chro\ R N R.IEEELGADAVYAGHNFR.N _C_eled:\celegans\Celegans\rep3_N15\chro\ R N K.FHGATSIQLVGDDLTVTNPK.R _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.VVLPVPAFNVINGGSHAGNK.L _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03726 transcription factor S-II status:partially_confirmed SW:P R U R.FGDMPQGTLDPEELAVQIEEK.L _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03737 locus:gst-13 glutathione S-transferase status:partially_confirm R U K.TAEESAMVDAFVDQFK.D _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE03770 status:partially_confirmed SW:Q23126 protein_id:caa R U R.IITPTLLSEYAYVLR.E _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.MFFLFPDPHFK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03794 cytosolic aminopeptidase status:confirmed SW:Q27245 prote R U K.LNQLGLLAIGNSDTSAR.L _CelegD:\celegans\Celegans\rep1\chro\ R U R.VQNVGDEFGQPIEISR.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U R.VAEYIQGAFANGNIK.V _Celegd:\celegans\Celegans\rep2\chro\ R U K.LNQLGLLAIGNSDTSAR.L _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE03812 locus:aco-1 iron-responsive element-binding like protein statu R U K.YLEASGLLPYLEK.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.SNLIGMGIIPFQYQAGQNADSLGLTGK.E _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.VGPITLHVPSGEELDIFDAAQK.Y _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U R.VILQDFTGVPAVVDLAAMR.D _Celegd:\celegans\Celegans\rep2\chro\ R U R.VILQDFTGVPAVVDLAAMR.D _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.YLEASGLLPYLEK.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R U K.VGPITLHVPSGEELDIFDAAQK.Y _Celegd:\celegans\Celegans\rep2_N15\chro\ S U R.VILQDFTGVPAVVDLAAMR.D _C_eled:\celegans\Celegans\rep3\chro\ R U K.VGPITLHVPSGEELDIFDAAQK.Y _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE03853 locus:msp-36 major sperm protein status:confirmed SW:P R N K.IVFNAPYDDK.H _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.IVFNAPYDDK.H _Celegd:\celegans\Celegans\rep2_N15\chro\
9 R N R.RIGYGIK.T _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE03880 protein disulfide-isomerase status:confirmed SW:Q11067 pro R U K.DGALPAEDDIDLSDIDLDKTEL _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.YFAPGSDVSDAQDYDGGR.Q _C_eled:\celegans\Celegans\rep3\chro\ P CE03921 locus:vit-5 status:partially_confirmed SW:P06125 protein_id:a S N R.VQSGIQMLQEIVK.K _Celegd:\celegans\Celegans\rep2\chro\ S N R.LVLDIVPTVATTHVTEMR.F _Celegd:\celegans\Celegans\rep2\chro\ S N K.SVQETLYPSEHIADLLIQLAQSPLSEK.Y _Celegd:\celegans\Celegans\rep2\chro\ P CE03926 status:confirmed SW:P48053 protein_id:aab R N K.YFTAPSATVIGVPNEELVDK.I _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE03957 S-adenosylmethionine synthetase status:partially_confirmed R N K.IVNDNFDLRPGMIIK.D _CelegD:\celegans\Celegans\rep1\chro\ R N R.SGELEWVRPDSK.T _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.IIVDTYGGWGAHGGGAFSGK.D _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N R.VHTVVISTQHSPDISLDDLR.K _CelegD:\celegans\Celegans\rep1_N15\chro1\ R N K.KPIYEPTAENGHFGHNEFPWEQPR.H _CelegD:\celegans\Celegans\rep1_N15\chro1\ S N K.TCNVLVALEQQSPEIAAGVHVDK.D _Celegd:\celegans\Celegans\rep2\chro\ R N K.IIVDTYGGWGAHGGGAFSGK.D _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.IVNDNFDLRPGMIIK.D _Celegd:\celegans\Celegans\rep2_N15\chro\ R N R.VHTVVISTQHSPDISLDDLR.K _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.KPIYEPTAENGHFGHNEFPWEQPR.H _Celegd:\celegans\Celegans\rep2_N15\chro\ P CE03959 S-adenosylmethionine synthetase status:partially_confirmed R N K.RPIYEPTAENGHFGHNNFPWEQPR.N _CelegD:\celegans\Celegans\rep1_N15\chro1\ S N R.VHTVVISTQHSPDISLDDLR.K _C_eled:\celegans\Celegans\rep3\chro\ S N K.TIYHLNPCGSFIIGGPMGDAGLTGR.K _C_eled:\celegans\Celegans\rep3\chro\ P CE03972 locus:pdi-2 protein disulfide isomerase status:confirmed SW: R N K.FKPDFEEITTENISK.F _CelegD:\celegans\Celegans\rep1\chro\ R U K.TFLEVAAGIDDVPFGISTEDAVK.S _CelegD:\celegans\Celegans\rep1\chro\ R N K.TGPVAKPLADADAVK.E _CelegD:\celegans\Celegans\rep1\chro\ R N R.DNTKNVLVEFYAPWCGHCK.Q _CelegD:\celegans\Celegans\rep1\chro\ R N R.DNTKNVLVEFYAPWCGHCK.Q _CelegD:\celegans\Celegans\rep1\chro\ R N R.LALVSEFTQETASVIFGGEIK.S _CelegD:\celegans\Celegans\rep1\chro\ R N R.LALVSEFTQETASVIFGGEIK.S _CelegD:\celegans\Celegans\rep1\chro\ R N K.SHNLLFVSK.E _CelegD:\celegans\Celegans\rep1\chro\ S N K.TGPVAKPLADADAVK.E _Celegd:\celegans\Celegans\rep2\chro\ S N K.SHNLLFVSK.E _Celegd:\celegans\Celegans\rep2\chro\ S N R.LALVSEFTQETASVIFGGEIK.S _Celegd:\celegans\Celegans\rep2\chro\ S N R.LALVSEFTQETASVIFGGEIK.S _Celegd:\celegans\Celegans\rep2\chro\ S N K.TGPVAKPLADADAVK.E _C_eled:\celegans\Celegans\rep3\chro\ S N K.NVLVEFYAPWCGHCK.Q _C_eled:\celegans\Celegans\rep3\chro\ S N R.DNTKNVLVEFYAPWCGHCK.Q _C_eled:\celegans\Celegans\rep3\chro\ S N R.LALVSEFTQETASVIFGGEIK.S _C_eled:\celegans\Celegans\rep3\chro\ P CE04010 ornithine aminotransferse precursor status:partially_confirme R N R.SIAAISASTDPDSFAR.F _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.AFYNNVLGEYEEYVTK.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ S N K.YNVLFITDEVQSGLGR.S _Celegd:\celegans\Celegans\rep2\chro\ R N K.LVENSAVMGDLLMSK.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.AFYNNVLGEYEEYVTK.L _Celegd:\celegans\Celegans\rep2_N15\chro\ R N K.KYNVLFITDEVQSGLGR.S _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.AFYNNVLGEYEEYVTK.L _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE04189 status:partially_confirmed TR:Q18577 protein_id:aaa S U K.ADEILDDQENTWNGANEK.L _C_eled:\celegans\Celegans\rep3\chro\ P CE04242 succinyl-coa synthetase beta chain status:partially_confirme R U K.HGCSVQNFVVASNR.K _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.HGCSVQNFVVASNR.K _Celegd:\celegans\Celegans\rep2\chro\ P CE04306 status:confirmed TR:Q19007 protein_id:aaa R U K.TYLLGFDPDAEAPLQFDGPHVIR.A _CelegD:\celegans\Celegans\rep1\chro\ R U K.IEFHFATNPYFK.N _CelegD:\celegans\Celegans\rep1\chro\ S U K.IEFHFATNPYFK.N _Celegd:\celegans\Celegans\rep2\chro\ R U K.ADSFFNFFEPPK.S _Celegd:\celegans\Celegans\rep2_N15\chro\ S U K.TYLLGFDPDAEAPLQFDGPHVIR.A _C_eled:\celegans\Celegans\rep3\chro\ R U K.EQIDTPILEGLEGDQLAELYK.A _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.ADSFFNFFEPPK.S _C_eled:\celegans\Celegans\rep3_N15\chro\ R U K.IEFHFATNPYFK.N _C_eled:\celegans\Celegans\rep3_N15\chro\ P CE04424 locus:vha-12 vacuolar ATP synthase (strong) status:confirme R U R.SVFESLDIGWQLLR.I _CelegD:\celegans\Celegans\rep1\chro\ R U K.IPIFSASGLPHNEIAAQIVR.Q _CelegD:\celegans\Celegans\rep1\chro\ R U R.LIYQTVCGVNGPLVILNDVK.F _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.LIYPPINVLPSLSR.L _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U R.SVFESLDIGWQLLR.I _CelegD:\celegans\Celegans\rep1_N15\chro1\ R U K.IPIFSASGLPHNEIAAQIVR.Q _CelegD:\celegans\Celegans\rep1_N15\chro1\ S U K.IPIFSASGLPHNEIAAQIVR.Q _Celegd:\celegans\Celegans\rep2\chro\ R U K.GPPVLAEDFLDINGQPINPWSR.I _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.EDHSDVSNQLYACYAIGK.D _Celegd:\celegans\Celegans\rep2_N15\chro\ R N R.GFPGYMYTDLATIYER.A _Celegd:\celegans\Celegans\rep2_N15\chro\ R N R.IYPEEMIQTGISAIDVMNSIAR.G _Celegd:\celegans\Celegans\rep2_N15\chro\ R U R.IALTSAEFLAYQCK.K _Celegd:\celegans\Celegans\rep2_N15\chro\
Supporting Information for Proteomics DOI /pmic
 Supporting Information for Proteomics DOI 10.1002/pmic.200401063 Linda L. Manza, Sheryl L. Stamer, Amy-Joan L. Ham, Simona G. Codreanu and Daniel C. Liebler Sample preparation and digestion for proteomic
Supporting Information for Proteomics DOI 10.1002/pmic.200401063 Linda L. Manza, Sheryl L. Stamer, Amy-Joan L. Ham, Simona G. Codreanu and Daniel C. Liebler Sample preparation and digestion for proteomic
Table 1s. Identified proteins along with modifications. Δ m/z Meta Range Sequence Modifications Accession Protein
 Table 1s. Identified proteins along with modifications Δ m/z Meta Range Sequence Modifications Accession Protein [ppm] Score -28.15 43.0 1256 - R.TLEDQVNELK.S 1265 6.52 27.5 11-32 R.AIQANINIPMGAFRPGAGQPPR.R
Table 1s. Identified proteins along with modifications Δ m/z Meta Range Sequence Modifications Accession Protein [ppm] Score -28.15 43.0 1256 - R.TLEDQVNELK.S 1265 6.52 27.5 11-32 R.AIQANINIPMGAFRPGAGQPPR.R
Supporting Information
 Supporting Information Expanding the dipeptidyl peptidase 4-regulated peptidome via an optimized peptidomics platform Arthur D. Tinoco, Debarati M. Tagore, and Alan Saghatelian* Department of Chemistry
Supporting Information Expanding the dipeptidyl peptidase 4-regulated peptidome via an optimized peptidomics platform Arthur D. Tinoco, Debarati M. Tagore, and Alan Saghatelian* Department of Chemistry
Exosomes Mediate Epithelium-Mesenchyme Crosstalk in Organ Development
 1 Exosomes Mediate Epithelium-Mesenchyme Crosstalk in Organ Development Nan Jiang 1,2, Lusai Xiang 2,5, Ling He 2,5, Guodong Yang 2, Jinxuan Zheng 2,5, Chenglin Wang 2,6, Yimei Zhang 3, Sainan Wang 2,
1 Exosomes Mediate Epithelium-Mesenchyme Crosstalk in Organ Development Nan Jiang 1,2, Lusai Xiang 2,5, Ling He 2,5, Guodong Yang 2, Jinxuan Zheng 2,5, Chenglin Wang 2,6, Yimei Zhang 3, Sainan Wang 2,
EKSAMEN I EMNE TBT4100 BIOKJEMI GRUNNKURS Tirsdag 14. desember 2006 Tid: kl. 09:00 14:00
 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI 1 Faglig kontakt under ekasmen: Institutt for bioteknologi, Gløshaugen Førsteamanuensis Sergey B. Zotchev, tlf. 98679. EKSAMEN
NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI 1 Faglig kontakt under ekasmen: Institutt for bioteknologi, Gløshaugen Førsteamanuensis Sergey B. Zotchev, tlf. 98679. EKSAMEN
Obligatorisk oppgave 2 MBV1030 Høst 2005
 Obligatorisk oppgave 2 MBV1030 Høst 2005 Levert av (navn): Første del: Flervalgsspørsmål. Angi det svaralternativet (ett) du mener er korrekt. I-1: Ved anaerob glykolyse dannes det laktat. Dersom glukosen
Obligatorisk oppgave 2 MBV1030 Høst 2005 Levert av (navn): Første del: Flervalgsspørsmål. Angi det svaralternativet (ett) du mener er korrekt. I-1: Ved anaerob glykolyse dannes det laktat. Dersom glukosen
Aminosyreomsetning og urea-syklus. Medisinstudiet semester 1A Asbjørn Nilsen, IKM
 Aminosyreomsetning og urea-syklus Medisinstudiet semester 1A Asbjørn Nilsen, IKM Aminosyreomsetning og ureasyklus Aminosyrer generelt Syntese av ikke-essensielle aminosyrer Nedbrytning av aminosyrer Utskilling
Aminosyreomsetning og urea-syklus Medisinstudiet semester 1A Asbjørn Nilsen, IKM Aminosyreomsetning og ureasyklus Aminosyrer generelt Syntese av ikke-essensielle aminosyrer Nedbrytning av aminosyrer Utskilling
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i MBV 1030 Generell biokjemi Eksamensdag: Mandag 6. desember 2004 Tid for eksamen: kl. 09.00 12.00 Oppgavesettet er på 9 sider Vedlegg:
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i MBV 1030 Generell biokjemi Eksamensdag: Mandag 6. desember 2004 Tid for eksamen: kl. 09.00 12.00 Oppgavesettet er på 9 sider Vedlegg:
FYS 3710 Biofysikk og Medisinsk Fysikk, Aminosyrer, Polypeptider, Proteiner
 FYS 3710 Biofysikk og Medisinsk Fysikk, 2016 5 Aminosyrer, Polypeptider, Proteiner Einar Sagstuen, Fysisk institutt, UiO 06.09.2016 1 sp n -hybridisering: for hovedkvantetall N=2 er de fire valensorbitalene
FYS 3710 Biofysikk og Medisinsk Fysikk, 2016 5 Aminosyrer, Polypeptider, Proteiner Einar Sagstuen, Fysisk institutt, UiO 06.09.2016 1 sp n -hybridisering: for hovedkvantetall N=2 er de fire valensorbitalene
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Midttermineksamen i: MBV3040 Nukleinsyrenes biokjemi Eksamensdag: 22. mars 2004 Tid for eksamen: 1,5 timer Oppgavesettet er på 10 sider.
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Midttermineksamen i: MBV3040 Nukleinsyrenes biokjemi Eksamensdag: 22. mars 2004 Tid for eksamen: 1,5 timer Oppgavesettet er på 10 sider.
BI 212- Protein Sorting - Kap. 17 Post-translasjonell modifisering og kvalitetskontroll i r-er (Del 17.6)
 Før de sekretoriske proteiner transporteres videre fra ER-lumen til sitt endelige bestemmelsessted, blir de modifisert ; Dannelse av disulfid-bindinger Foldinger av proteinet Påleiring og prosessering
Før de sekretoriske proteiner transporteres videre fra ER-lumen til sitt endelige bestemmelsessted, blir de modifisert ; Dannelse av disulfid-bindinger Foldinger av proteinet Påleiring og prosessering
Forelesninger i BI Cellebiologi. Denaturering og renaturering. Figure 3-13
 Figure 3.9 Denaturering og renaturering Figure 3-13 Denaturering og renaturering Figure 3-14 Viser tre trinn i refolding av et protein som har vært denaturert. Molten globule -formen er en intermediær
Figure 3.9 Denaturering og renaturering Figure 3-13 Denaturering og renaturering Figure 3-14 Viser tre trinn i refolding av et protein som har vært denaturert. Molten globule -formen er en intermediær
BI 212- Protein Sorting - Kap. 17 Syntese og mål for mitokondrie- og kloroplast-proteiner (forts.)
 Syntese og mål for mitokondrie- og kloroplast-proteiner (forts.) Veiene for opptak fra cytosol av kloroplast-proteiner Opptak av proteiner fra cytosol til kloroplaster ligner mye på mitokondrie-importen
Syntese og mål for mitokondrie- og kloroplast-proteiner (forts.) Veiene for opptak fra cytosol av kloroplast-proteiner Opptak av proteiner fra cytosol til kloroplaster ligner mye på mitokondrie-importen
EKSAMEN I EMNE TBT4102 BIOKJEMI I. 2. desember 2011 kl
 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Hanne Jørgensen, tlf. 591685 EKSAMEN I EMNE TBT4102 BIOKJEMI
NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Hanne Jørgensen, tlf. 591685 EKSAMEN I EMNE TBT4102 BIOKJEMI
EKSAMEN I EMNE TBT4102 BIOKJEMI I. 3. desember 2012 kl
 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Kjell M. Vårum, tlf. 93022165 EKSAMEN I EMNE TBT4102 BIOKJEMI
NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Kjell M. Vårum, tlf. 93022165 EKSAMEN I EMNE TBT4102 BIOKJEMI
Cellular Energetics- Kap. 16
 Forelesninger i BI 212 - Cellebiologi - Våren 2002 Cellular Energetics- Kap. 16 Tor-Henning Iversen, Plantebiosenteret (PBS),Botanisk institutt,ntnu e-mail : Tor-Henning.Iversen@chembio chembio.ntnu.no
Forelesninger i BI 212 - Cellebiologi - Våren 2002 Cellular Energetics- Kap. 16 Tor-Henning Iversen, Plantebiosenteret (PBS),Botanisk institutt,ntnu e-mail : Tor-Henning.Iversen@chembio chembio.ntnu.no
LEHNINGER PRINCIPLES OF BIOCHEMISTRY
 David L. Nelson and Michael M. Cox LEHNINGER PRINCIPLES OF BIOCHEMISTRY Fifth Edition CHAPTER 19 Oxidative Phosphorylation 2008 W. H. Freeman and Company Cellulær respirasjon: siste trinn Elektronoverføring
David L. Nelson and Michael M. Cox LEHNINGER PRINCIPLES OF BIOCHEMISTRY Fifth Edition CHAPTER 19 Oxidative Phosphorylation 2008 W. H. Freeman and Company Cellulær respirasjon: siste trinn Elektronoverføring
Table S1. Phosphopeptides regulated by SSE and insulin.
 Table S. Phosphopeptides regulated by SSE and insulin. Phospho Site AAADDG EEPKS(ph) EPETK AAADDG EEPKS(ph) EPETKK AEDGAA PS(ph)PSSE T(ph)PKKK AEDGAA PSPSSET(p h)pkk AEGSLHS S(ph)PAGP SSSK AENQRP AEDSALS(
Table S. Phosphopeptides regulated by SSE and insulin. Phospho Site AAADDG EEPKS(ph) EPETK AAADDG EEPKS(ph) EPETKK AEDGAA PS(ph)PSSE T(ph)PKKK AEDGAA PSPSSET(p h)pkk AEGSLHS S(ph)PAGP SSSK AENQRP AEDSALS(
Fastere filet - Aktivitet 2
 Fastere filet - Aktivitet 2 MRI, protease og protein analyser Iciar Martinez 1,2, Rasa Slizyté 1, Pål Anders Wang 2,Stine W. Dahle 1, Michiaki Yamashita 3, Ragnar Olsen 2, Emil Veliuylin 1 og Ulf Erikson
Fastere filet - Aktivitet 2 MRI, protease og protein analyser Iciar Martinez 1,2, Rasa Slizyté 1, Pål Anders Wang 2,Stine W. Dahle 1, Michiaki Yamashita 3, Ragnar Olsen 2, Emil Veliuylin 1 og Ulf Erikson
Regularitati ascunse si corelatii in nano-bio-structuri
 NATIONAL INSTITUTE OF MATERIALS PHYSICS BUCHAREST-MAGURELE Atomistilor Str. bis, P.O. Box MG-7, 77 Magurele-Ilfov, Romania Phone: +() 98, Fax: +() 977, email: pintilie@infim.ro, http://www.infim.ro Regularitati
NATIONAL INSTITUTE OF MATERIALS PHYSICS BUCHAREST-MAGURELE Atomistilor Str. bis, P.O. Box MG-7, 77 Magurele-Ilfov, Romania Phone: +() 98, Fax: +() 977, email: pintilie@infim.ro, http://www.infim.ro Regularitati
Regulering av DNA Transkripsjon i Eukaryote Organismer. ID, Kull 99, Vår 2001 Frank Skorpen IKM, DMF
 Regulering av DNA Transkripsjon i Eukaryote Organismer ID, Kull 99, Vår 2001 Frank Skorpen IKM, DMF 1 Regulering av gen-uttrykk på mange nivåer Klargjøring av DNA Transkripsjon Initiering Stopp hnrna prosessering
Regulering av DNA Transkripsjon i Eukaryote Organismer ID, Kull 99, Vår 2001 Frank Skorpen IKM, DMF 1 Regulering av gen-uttrykk på mange nivåer Klargjøring av DNA Transkripsjon Initiering Stopp hnrna prosessering
Accession start end. Bifunctional purine biosynthesis protein ADE17 [Includes: Phosphoribosylaminoimidazolecarboxamide formyltransferase
 Table S1A. List of 379 unique N-terminal peptides (start position 1 or 2) identified in the proteome of the control yeast strain ( 12 C6 ), the ynata deficient strain expressing hnata ( 13 C6) and/or the
Table S1A. List of 379 unique N-terminal peptides (start position 1 or 2) identified in the proteome of the control yeast strain ( 12 C6 ), the ynata deficient strain expressing hnata ( 13 C6) and/or the
l-l oco UNIVERSITETET IOSLO Det matematisk-naturvitenskapelige fakuftet fi t
 UNIVERSITETET IOSLO Det matematisk-naturvitenskapelige fakuftet Eksamen i: MBV1030 Genercll biokjemi Eksamensdag: S. desember 2006 Tid for eksamen: 15.30 - {9.30 Oppgavesettet er pi 7 side(r) Vedlegg:
UNIVERSITETET IOSLO Det matematisk-naturvitenskapelige fakuftet Eksamen i: MBV1030 Genercll biokjemi Eksamensdag: S. desember 2006 Tid for eksamen: 15.30 - {9.30 Oppgavesettet er pi 7 side(r) Vedlegg:
Idrett og energiomsetning
 1 Medisin stadium IA, Tonje S. Steigedal 2 ATP er den eneste forbindelsen som kan drive kontraksjon av musklene. ATPnivået i muskelcellene er imidlertid begrenset, og må etterfylles kontinuerlig. Ved ulike
1 Medisin stadium IA, Tonje S. Steigedal 2 ATP er den eneste forbindelsen som kan drive kontraksjon av musklene. ATPnivået i muskelcellene er imidlertid begrenset, og må etterfylles kontinuerlig. Ved ulike
UNIVERSITETET I OSLO. Det matematisk-naturvitenskapelige fakultet
 UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i MBV 1030 Generell biokjemi Eksamensdag: 6. /7. januar 2005 Tid for eksamen: Oppgavesettet er på 6 sider Vedlegg: 1 Tillatte hjelpemidler:
UNIVERSITETET I OSLO Det matematisk-naturvitenskapelige fakultet Eksamen i MBV 1030 Generell biokjemi Eksamensdag: 6. /7. januar 2005 Tid for eksamen: Oppgavesettet er på 6 sider Vedlegg: 1 Tillatte hjelpemidler:
Proteiner og aminosyrer
 Proteiner og aminosyrer Presentasjonsplan 1/2 Cellen Grunnleggende komponenter DNA til mrna til proteiner Den genetiske koden: Hva er et codon? Presentasjonsplan 2/2 Aminosyrer del 1 Hvilke molekyler er
Proteiner og aminosyrer Presentasjonsplan 1/2 Cellen Grunnleggende komponenter DNA til mrna til proteiner Den genetiske koden: Hva er et codon? Presentasjonsplan 2/2 Aminosyrer del 1 Hvilke molekyler er
Hvordan holde styr på proteasene?? Harald; 31.01.08
 Hvordan holde styr på proteasene?? Harald; 31.01.08 Protease? Proteinase? Spalter proteiner Peptidase? Spalter peptider? Spalter peptidbindinger Exo-peptidaser Endo-peptidaser Endo Ekso- Proteaser er enzymer!
Hvordan holde styr på proteasene?? Harald; 31.01.08 Protease? Proteinase? Spalter proteiner Peptidase? Spalter peptider? Spalter peptidbindinger Exo-peptidaser Endo-peptidaser Endo Ekso- Proteaser er enzymer!
Klassifisering av enzymer. Litt historikk. Generell mekanisme for enzymkatalyse:
 Enzymologi De aller fleste enzymer består helt eller delvis av proteiner (unntak: ribozymer) Enzymer har evnen til å katalysere kjemiske reaksjoner i kroppen ved lav temperatur. Selv kjemiske reaksjoner
Enzymologi De aller fleste enzymer består helt eller delvis av proteiner (unntak: ribozymer) Enzymer har evnen til å katalysere kjemiske reaksjoner i kroppen ved lav temperatur. Selv kjemiske reaksjoner
EKSAMEN I EMNE SIF4070 CELLEBIOLOGI
 Side 1 av 7 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE SIF4070 CELLEBIOLOGI Faglig kontakt under eksamen: professor Catharina Davies Tel 73593688 eller 41666231
Side 1 av 7 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE SIF4070 CELLEBIOLOGI Faglig kontakt under eksamen: professor Catharina Davies Tel 73593688 eller 41666231
Forelesninger i BI Cellebiologi. Enzymer : senker aktiveringsenergien. Figure 6.13
 Enzymer : senker aktiveringsenergien Figure 6.13 Aktive seter : camp-avhengig protein kinase *For å illustrere hvordan det aktive setet binder et spesifikt substrat er valgt som eksempel camp-avhengig
Enzymer : senker aktiveringsenergien Figure 6.13 Aktive seter : camp-avhengig protein kinase *For å illustrere hvordan det aktive setet binder et spesifikt substrat er valgt som eksempel camp-avhengig
REGULERING AV TRANSKRIPSJON I EUKARYOTE ORGANISMER
 REGULERING AV TRANSKRIPSJON I EUKARYOTE ORGANISMER av Frank Skorpen, IKM, DMF, NTNU FORELESNING April 2001 Cellebiologi - Hovedfag 1 REGULERING AV TRANSKRIPSJON I EUKARYOTE ORGANISMER Det er flere nøkkel-mekanismer
REGULERING AV TRANSKRIPSJON I EUKARYOTE ORGANISMER av Frank Skorpen, IKM, DMF, NTNU FORELESNING April 2001 Cellebiologi - Hovedfag 1 REGULERING AV TRANSKRIPSJON I EUKARYOTE ORGANISMER Det er flere nøkkel-mekanismer
De aller fleste enzymer består helt eller delvis av proteiner (unntak: ribozymer) Enzymer har evnen til å katalysere kjemiske reaksjoner i kroppen
 Enzymologi De aller fleste enzymer består helt eller delvis av proteiner (unntak: ribozymer) Enzymer har evnen til å katalysere kjemiske reaksjoner i kroppen ved lav temperatur. Selv kjemiske reaksjoner
Enzymologi De aller fleste enzymer består helt eller delvis av proteiner (unntak: ribozymer) Enzymer har evnen til å katalysere kjemiske reaksjoner i kroppen ved lav temperatur. Selv kjemiske reaksjoner
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Side 1 Det matematisk-naturvitenskapelige fakultet Bokmål Eksamen i MBV 3010, Videregående cellebiologi. Eksamensdag: august 2006. Tid for eksamen: kl (3 timer). Oppgavesettet er på
UNIVERSITETET I OSLO Side 1 Det matematisk-naturvitenskapelige fakultet Bokmål Eksamen i MBV 3010, Videregående cellebiologi. Eksamensdag: august 2006. Tid for eksamen: kl (3 timer). Oppgavesettet er på
UNIVERSITETET I OSLO
 UNIVERSITETET I OSLO Side 1 Det matematisk-naturvitenskapelige fakultet Eksamen i MBV 3010, Videregående cellebiologi. Eksamensdag: 14. juni 2006. Tid for eksamen: kl 14.30 (3 timer). Oppgavesettet er
UNIVERSITETET I OSLO Side 1 Det matematisk-naturvitenskapelige fakultet Eksamen i MBV 3010, Videregående cellebiologi. Eksamensdag: 14. juni 2006. Tid for eksamen: kl 14.30 (3 timer). Oppgavesettet er
Mikrofôr til marine fiskelarver Kan man unngå lekkasje?
 Mikrofôr til marine fiskelarver Kan man unngå lekkasje? IMR IMR Andreas Nordgreen 14.02.2012 1 Hvordan levere vannløselige næringsstoff til saktespisende arter Baer 14.02.2012 2 Nødvendig første periode
Mikrofôr til marine fiskelarver Kan man unngå lekkasje? IMR IMR Andreas Nordgreen 14.02.2012 1 Hvordan levere vannløselige næringsstoff til saktespisende arter Baer 14.02.2012 2 Nødvendig første periode
Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
 Side 1 av 12 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
Side 1 av 12 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
Pyruvat dehydrogenase er et multienzymkompleks. Oksydativ nebrytning av pyrodruesyre skjer i mitokondriene
 Medisin, stadium 1A, Geir Slupphaug, IKM Sitronsyresyklus Når cellene har tilstrekkelig tilgang på oksygen, vil ikke pyruvat dannet i glykolysen omdannes til laktat, men vil i stedet omdannes til Acetyl-
Medisin, stadium 1A, Geir Slupphaug, IKM Sitronsyresyklus Når cellene har tilstrekkelig tilgang på oksygen, vil ikke pyruvat dannet i glykolysen omdannes til laktat, men vil i stedet omdannes til Acetyl-
Proteiner og proteinstrukturer
 Medisin stadium 1A Geir Slupphaug, IKM Proteiner og proteinstrukturer Alle proteiner er bygd opp av aminosyrer. Dette er en gruppe organiske molekyler som inneholder både en karboksylsyregruppe (-COOH)
Medisin stadium 1A Geir Slupphaug, IKM Proteiner og proteinstrukturer Alle proteiner er bygd opp av aminosyrer. Dette er en gruppe organiske molekyler som inneholder både en karboksylsyregruppe (-COOH)
Enzymer og enzymkinetikk
 1, IA 2013 2 Relevante læringsmål, Biokjemi: 2.1.1.17 forklare enzymers hovedfunksjon, og definere begrepene K m og V max 2.1.1.18 forklare hvordan enzymers aktivitet kan reguleres i cellene Relevant studiemateriell:
1, IA 2013 2 Relevante læringsmål, Biokjemi: 2.1.1.17 forklare enzymers hovedfunksjon, og definere begrepene K m og V max 2.1.1.18 forklare hvordan enzymers aktivitet kan reguleres i cellene Relevant studiemateriell:
Bioenergetikk og Krebs syklus Oksidativ fosforylering
 Bioenergetikk og Krebs syklus Oksidativ fosforylering Bioenergetikk, IA 2015 Det store bildet Bioenergetikk ATP Den mengden ATP som brytes ned og dannes pr dag hos mennesket, tilsvarer omtrent kroppsvekten
Bioenergetikk og Krebs syklus Oksidativ fosforylering Bioenergetikk, IA 2015 Det store bildet Bioenergetikk ATP Den mengden ATP som brytes ned og dannes pr dag hos mennesket, tilsvarer omtrent kroppsvekten
Sammendrag. Sammendrag
 Forord Forord Denne masteroppgaven ble utført høsten 213, samt deler av våren 214, ved Institutt for kjemi, bioteknologi og matvitenskap, Norges miljø og biovitenskapelige universitet. Først og fremst
Forord Forord Denne masteroppgaven ble utført høsten 213, samt deler av våren 214, ved Institutt for kjemi, bioteknologi og matvitenskap, Norges miljø og biovitenskapelige universitet. Først og fremst
Proteiner og aminosyrer
 Proteiner og aminosyrer Presentasjonsplan 1/2 Introduksjon DNA mrna pep=d Den gene=ske koden: Hva er et codon? Aminosyrer del 1 Hvilke molekyler er dece? (Klassifisering) Kilder og syntese Presentasjonsplan
Proteiner og aminosyrer Presentasjonsplan 1/2 Introduksjon DNA mrna pep=d Den gene=ske koden: Hva er et codon? Aminosyrer del 1 Hvilke molekyler er dece? (Klassifisering) Kilder og syntese Presentasjonsplan
Transkripsjon v-02 9 Gjør rede for generelle prinsipper ved transkripsjonsregulering i eukaryote celler.
 Transkripsjon v-02 9 Gjør rede for generelle prinsipper ved transkripsjonsregulering i eukaryote celler. Først må DNA gjøres klart til transkripsjon. Dette kan innebære prosesser som nucleosom "unfolding",
Transkripsjon v-02 9 Gjør rede for generelle prinsipper ved transkripsjonsregulering i eukaryote celler. Først må DNA gjøres klart til transkripsjon. Dette kan innebære prosesser som nucleosom "unfolding",
Ernæringsterapi. Aminosyrer. Aminosyrer. Aminosyrer (2) Aminosyrer (3) Signalstoffer. Neurotransmittere- signalstoffer. Jens Veiersted 12.08.
 Byggestener i kroppen Stort organisk molekyl Inneholder En syre gruppe COOH En amino gruppe NH 2 Er alltid et hvitt krystallinsk pulver Løses i vann Har mange oppgaver i kroppen Alle 8 essensielle må være
Byggestener i kroppen Stort organisk molekyl Inneholder En syre gruppe COOH En amino gruppe NH 2 Er alltid et hvitt krystallinsk pulver Løses i vann Har mange oppgaver i kroppen Alle 8 essensielle må være
Cytoskjelettet. plasmamembran. Terje Espevik IKM. plasmamembran. Oversikt over Aktinfilamenter Mikrotubuli Intermediærfilamenter
 plasmamembran Cytoskjelettet Terje Espevik IKM plasmamembran Oversikt over Aktinfilamenter Mikrotubuli Intermediærfilamenter Intermediærfilamenter; inndeling Aktinfilamenter Struktur av G- og F-aktin Funksjoner
plasmamembran Cytoskjelettet Terje Espevik IKM plasmamembran Oversikt over Aktinfilamenter Mikrotubuli Intermediærfilamenter Intermediærfilamenter; inndeling Aktinfilamenter Struktur av G- og F-aktin Funksjoner
Uønsket opptak av store molekyler i lever et problem som kan overkommes?
 Uønsket opptak av store molekyler i lever et problem som kan overkommes? Bård Smedsrød Founder and CSO Blood clearance function of the liver: Fate of i.v. administered macromolecule ( 125 I-FSA) Blood
Uønsket opptak av store molekyler i lever et problem som kan overkommes? Bård Smedsrød Founder and CSO Blood clearance function of the liver: Fate of i.v. administered macromolecule ( 125 I-FSA) Blood
Kapittel 14: Det eukaryote genom og dets uttrykksregulering
 Kapittel 14: Det eukaryote genom og dets uttrykksregulering Innhold: 1. Det humane genom 2. Struktur av protein-kodende gener 3. RNA processering 4. Transkripsjonell kontroll 5. Posttranskripsjonell kontroll
Kapittel 14: Det eukaryote genom og dets uttrykksregulering Innhold: 1. Det humane genom 2. Struktur av protein-kodende gener 3. RNA processering 4. Transkripsjonell kontroll 5. Posttranskripsjonell kontroll
Cellesyklus. Medisin stadium IA, 17. september 2012
 Cellesyklus Medisin stadium IA, 17. september 2012 Trude Helen Flo Cellesyklus: En oversikt Definisjoner De ulike fasene av cellesyklus Regulering av cellesyklus Kort om apoptose Kort om stamceller Cellesyklus:
Cellesyklus Medisin stadium IA, 17. september 2012 Trude Helen Flo Cellesyklus: En oversikt Definisjoner De ulike fasene av cellesyklus Regulering av cellesyklus Kort om apoptose Kort om stamceller Cellesyklus:
Protein Sorting- Kap. 17
 Forelesninger i BI 212 - Cellebiologi - Våren 2002 Protein Sorting- Kap. 17 Tor-Henning Iversen, Plantebiosenteret (PBS),Botanisk institutt,ntnu e-mail : Tor- Henning.Iversen@chembio chembio.ntnu.no Tlf.
Forelesninger i BI 212 - Cellebiologi - Våren 2002 Protein Sorting- Kap. 17 Tor-Henning Iversen, Plantebiosenteret (PBS),Botanisk institutt,ntnu e-mail : Tor- Henning.Iversen@chembio chembio.ntnu.no Tlf.
Anne Helene Barsnes 03.06.2011
 TBT4170 Terms Begreper og biologiske prosesser i Bioteknologi Anne Helene Barsnes 03.06.2011 Innholdsfortegnelse 1. Tree of life/cell biology/basic chemistry of life... 4 Livets tre... 4 2. Enzymes...
TBT4170 Terms Begreper og biologiske prosesser i Bioteknologi Anne Helene Barsnes 03.06.2011 Innholdsfortegnelse 1. Tree of life/cell biology/basic chemistry of life... 4 Livets tre... 4 2. Enzymes...
Kapittel 12: FRA DNA TIL PROTEIN:
 Kapittel 12: FRA DNA TIL PROTEIN: fra genotype til fenotype 1. Gener og polypeptider 2. DNA, RNA og informasjonsflow 3. Transkripsjon: DNA-dirigert RNA-syntese 4. Den genetiske kode 5. Aktører i Translasjon
Kapittel 12: FRA DNA TIL PROTEIN: fra genotype til fenotype 1. Gener og polypeptider 2. DNA, RNA og informasjonsflow 3. Transkripsjon: DNA-dirigert RNA-syntese 4. Den genetiske kode 5. Aktører i Translasjon
Konjugeringsenzymer. Glukuronidering Sulfatering Acetylering Metylering Glutationkonjugering Aminosyrekonjugering
 Konjugeringsenzymer Glukuronidering Sulfatering Acetylering Metylering Glutationkonjugering Aminosyrekonjugering Konjugeringsenzymer Konjugat Kosubstrat Enzym Glukosid UDP-G Glukosyltransferase Glukuronid
Konjugeringsenzymer Glukuronidering Sulfatering Acetylering Metylering Glutationkonjugering Aminosyrekonjugering Konjugeringsenzymer Konjugat Kosubstrat Enzym Glukosid UDP-G Glukosyltransferase Glukuronid
Fra lav-verdi biomasse til høy-verdi fôrressurs hvilke muligheter finnes? Margareth Øverland Aquaculture Protein Centre
 Fra lav-verdi biomasse til høy-verdi fôrressurs hvilke muligheter finnes? Margareth Øverland Aquaculture Protein Centre Produksjon av laksefisk i Norge og globalt 2,5 Produksjon og verdi av laks og regnbueørret
Fra lav-verdi biomasse til høy-verdi fôrressurs hvilke muligheter finnes? Margareth Øverland Aquaculture Protein Centre Produksjon av laksefisk i Norge og globalt 2,5 Produksjon og verdi av laks og regnbueørret
Hva viser genanalyser av muskulatur hos laks med mørke flekker. Aleksei Krasnov, Hooman Moghadam Nofima, Ås
 Hva viser genanalyser av muskulatur hos laks med mørke flekker Aleksei Krasnov, Hooman Moghadam Nofima, Ås Spørsmål Har mørke flekker lik eller ulik profil? Vevsskade og nydannelse av vev? Type betennelse?
Hva viser genanalyser av muskulatur hos laks med mørke flekker Aleksei Krasnov, Hooman Moghadam Nofima, Ås Spørsmål Har mørke flekker lik eller ulik profil? Vevsskade og nydannelse av vev? Type betennelse?
Besvarelse SIF4070 Cellebiologi 31. mai 2002
 1 Besvarelse SIF4070 Cellebiologi 31. mai 2002 Oppgave 1: Syntese av plasmamembranen. Transport over plasmamembranen a) Proteinet når ER Transport til ER membranen Transmembranproteiner som skal til ER
1 Besvarelse SIF4070 Cellebiologi 31. mai 2002 Oppgave 1: Syntese av plasmamembranen. Transport over plasmamembranen a) Proteinet når ER Transport til ER membranen Transmembranproteiner som skal til ER
Medisin, stadium 1A, Geir Slupphaug, IKM Sitronsyresyklus
 Medisin, stadium 1A, Geir Slupphaug, IKM Sitronsyresyklus Når cellene har tilstrekkelig tilgang på oksygen, vil ikke pyruvat dannet i glykolysen omdannes til laktat, men vil i stedet omdannes til Acetyl-
Medisin, stadium 1A, Geir Slupphaug, IKM Sitronsyresyklus Når cellene har tilstrekkelig tilgang på oksygen, vil ikke pyruvat dannet i glykolysen omdannes til laktat, men vil i stedet omdannes til Acetyl-
Biokjemiske reaksjonsruter
 Forelesning i glykolyse. Medisin stadium 1A, Geir Slupphaug, IKM Biokjemiske reaksjonsruter Kan virke svært forvirrende ved første øyekast. Slike oversikter er imidlertid ikke annet enn en form for kart,
Forelesning i glykolyse. Medisin stadium 1A, Geir Slupphaug, IKM Biokjemiske reaksjonsruter Kan virke svært forvirrende ved første øyekast. Slike oversikter er imidlertid ikke annet enn en form for kart,
Proteiner og proteinstrukturer
 Medisin stadium 1A Geir Slupphaug, IKM Proteiner og proteinstrukturer Alle proteiner er bygd opp av aminosyrer. Dette er en gruppe organiske molekyler som inneholder både en karboksylsyregruppe (-) og
Medisin stadium 1A Geir Slupphaug, IKM Proteiner og proteinstrukturer Alle proteiner er bygd opp av aminosyrer. Dette er en gruppe organiske molekyler som inneholder både en karboksylsyregruppe (-) og
Oppgave 4: Fermentering av karbohydrater og identifisering av disakkaridhydrolaser i gjær
 TBT4107 Biokjemi 2 Oppgave 4: Fermentering av karbohydrater og identifisering av disakkaridhydrolaser i gjær Gruppe 20 Katrine Bringe, Lene Brattsti Dypås og Ove Øyås NTNU, 15. mars 2012 Innhold Sammendrag
TBT4107 Biokjemi 2 Oppgave 4: Fermentering av karbohydrater og identifisering av disakkaridhydrolaser i gjær Gruppe 20 Katrine Bringe, Lene Brattsti Dypås og Ove Øyås NTNU, 15. mars 2012 Innhold Sammendrag
Figurer og diagrammer
 Figurer og diagrammer Aminor Stort organisk molekyl En gruppe COOH En amino gruppe NH Et hvitt krystallinsk pulver Løses i vann Har mange oppgaver i kroppen Alle 8 essensielle må være til stede for å bygge
Figurer og diagrammer Aminor Stort organisk molekyl En gruppe COOH En amino gruppe NH Et hvitt krystallinsk pulver Løses i vann Har mange oppgaver i kroppen Alle 8 essensielle må være til stede for å bygge
Oppgavesettet utgjør totalt 100 vekttall. Antall vekttall er vist i parentes foran hver spørsmålsgruppe.
 Kontinuasjonseksamen, MEDSEM/ODSEM/ERNSEM2 høst 2009 Onsdag 17. februar 2010 kl. 09:00-15:00 Oppgavesettet består av 4 sider Hjelpemidler: Norsk rettskrivningsordbok Oppgavesettet utgjør totalt 100 vekttall.
Kontinuasjonseksamen, MEDSEM/ODSEM/ERNSEM2 høst 2009 Onsdag 17. februar 2010 kl. 09:00-15:00 Oppgavesettet består av 4 sider Hjelpemidler: Norsk rettskrivningsordbok Oppgavesettet utgjør totalt 100 vekttall.
Amplifikasjonsteknikker - andre metoder
 Amplifikasjonsteknikker - andre metoder Svein Arne Nordbø TH-28973 17.03.15 Alternative amplifikasjonsmetoder Templat-amplifikasjons metoder Signal-amplifikasjonsmetoder Templat-amplifikasjons metoder
Amplifikasjonsteknikker - andre metoder Svein Arne Nordbø TH-28973 17.03.15 Alternative amplifikasjonsmetoder Templat-amplifikasjons metoder Signal-amplifikasjonsmetoder Templat-amplifikasjons metoder
Aminosyresammensetning i korn
 230 M. Åssveen / Grønn kunnskap 9 (2) Aminosyresammensetning i korn Mauritz Åssveen / mauritz.aassveen@planteforsk.no Planteforsk Apelsvoll forskingssenter Bakgrunn Den overveiende delen av bygg og havre
230 M. Åssveen / Grønn kunnskap 9 (2) Aminosyresammensetning i korn Mauritz Åssveen / mauritz.aassveen@planteforsk.no Planteforsk Apelsvoll forskingssenter Bakgrunn Den overveiende delen av bygg og havre
EKSAMEN I BI1001 CELLE OG MOLEKYLÆRBIOLOGI
 Fag/ Emnekode: BI1001 Dato: Kandidatnr.: Norges Teknisk Naturvitenskapelige Universitet Institutt for Biologi EKSAMEN I BI1001 CELLE OG MOLEKYLÆRBIOLOGI Ansvarlig kontakt ved eksamen: Berit Johansen Phone:
Fag/ Emnekode: BI1001 Dato: Kandidatnr.: Norges Teknisk Naturvitenskapelige Universitet Institutt for Biologi EKSAMEN I BI1001 CELLE OG MOLEKYLÆRBIOLOGI Ansvarlig kontakt ved eksamen: Berit Johansen Phone:
Universitetet i Oslo
 Universitetet i Oslo Det matematisk-naturvitenskapelige fakultet Eksamen i: MBV1030 - Generell biokjemi Eksamenstype: Midtterminseksamen Eksamensdag: Mandag 11. oktober 2004 Tid for eksamen: kl 14.30 17.30
Universitetet i Oslo Det matematisk-naturvitenskapelige fakultet Eksamen i: MBV1030 - Generell biokjemi Eksamenstype: Midtterminseksamen Eksamensdag: Mandag 11. oktober 2004 Tid for eksamen: kl 14.30 17.30
Verdens mest kjente Biolog var og Geolog
 Geologi Biologi Verdens mest kjente Biolog var og Geolog Earth System Science Kartlegging av Dyphavene Gir ny Forståelse Kartlegging av havbunnen med Multistråle Ekkolodd 600 m 3500 m 10km EM-100 75
Geologi Biologi Verdens mest kjente Biolog var og Geolog Earth System Science Kartlegging av Dyphavene Gir ny Forståelse Kartlegging av havbunnen med Multistråle Ekkolodd 600 m 3500 m 10km EM-100 75
Modeling the immune system
 Master Modeling the immune system nnate mmunity Innate immunity or natural immunity refers to body's defense mechanisms that: are available instantaneously and for the first few day after microbial infection.
Master Modeling the immune system nnate mmunity Innate immunity or natural immunity refers to body's defense mechanisms that: are available instantaneously and for the first few day after microbial infection.
Glufosinat-ammonium. DAS mepsps
 Funksjonen til pat-genet er å danne glufosinat-ammonium-toleranse i DAS-81419-2 x DAS44406-6 planten og danner immunitet mot kontakt med ugressmiddel med glufosinatammonium. Det innsatte pat-genet er for
Funksjonen til pat-genet er å danne glufosinat-ammonium-toleranse i DAS-81419-2 x DAS44406-6 planten og danner immunitet mot kontakt med ugressmiddel med glufosinatammonium. Det innsatte pat-genet er for
Intracellulær proteinsortering, endocytose, eksocytose.. riktig protein på riktig sted i cellen.
 Intracellulær proteinsortering, endocytose, eksocytose.. riktig protein på riktig sted i cellen. Forelesning for IB, mai 2008 Terje Espevik, IKM "What the?... This is lemonade! Where's my culture of amoebic
Intracellulær proteinsortering, endocytose, eksocytose.. riktig protein på riktig sted i cellen. Forelesning for IB, mai 2008 Terje Espevik, IKM "What the?... This is lemonade! Where's my culture of amoebic
Hyperammonemi- medfødte stoffskiftesykdommer. Trine Tangeraas Sept 2017
 Hyperammonemi- medfødte stoffskiftesykdommer Trine Tangeraas Sept 2017 To hovedbudskap : 1. Mål ammoniakk hos alle med encefalopati og alle nyfødte med sepsismistanke 2. Start behandling-unngå forsinkelse-mye
Hyperammonemi- medfødte stoffskiftesykdommer Trine Tangeraas Sept 2017 To hovedbudskap : 1. Mål ammoniakk hos alle med encefalopati og alle nyfødte med sepsismistanke 2. Start behandling-unngå forsinkelse-mye
NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE TFY4260 CELLEBIOLOGI OG CELLULÆR BIOFYSIKK
 Side av 1 av 4 Studentnr... Studieprogram... Sidenr. NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE TFY4260 CELLEBIOLOGI OG CELLULÆR BIOFYSIKK Faglig kontakt under eksamen:
Side av 1 av 4 Studentnr... Studieprogram... Sidenr. NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE TFY4260 CELLEBIOLOGI OG CELLULÆR BIOFYSIKK Faglig kontakt under eksamen:
Advances in selectable marker genes for plant transformation
 Journal of Plant Physiology 165 (2008) 1698 1716 www.elsevier.de/jplph Advances in selectable marker genes for plant transformation Isaac Kirubakaran Sundar, Natarajan Sakthivel Department of Biotechnology,
Journal of Plant Physiology 165 (2008) 1698 1716 www.elsevier.de/jplph Advances in selectable marker genes for plant transformation Isaac Kirubakaran Sundar, Natarajan Sakthivel Department of Biotechnology,
Oncogenic Mutations Affecting Cell Proliferation
 Oncogenic Mutations Affecting Cell Proliferation Fra RTK til Nucleus (Boka s.1070-74) Normalt kreves et vekst stimulerende signal ( growth factor eks. PDGF, EGF, NGF) for at celler skal gå inn i celledeling,
Oncogenic Mutations Affecting Cell Proliferation Fra RTK til Nucleus (Boka s.1070-74) Normalt kreves et vekst stimulerende signal ( growth factor eks. PDGF, EGF, NGF) for at celler skal gå inn i celledeling,
Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
 Side 1 av 13 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
Side 1 av 13 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
C rhinovirus picornain 3C A HIV-1 retropepsin A feline immunodeficiency virus retropepsin
 Peptide Sequence Annotation AADHDG CAS-L1 AAEAISDA AAEHDG CAS-L2 AAEYGAEA AAGAMFLE M10.007-stromelysin 3 (MMP-11) AAQNASMW A06.001-nodavirus endopeptidase AASGFASP M04.003-vibriolysin ADAHDG CAS-L3 ADAPKGGG
Peptide Sequence Annotation AADHDG CAS-L1 AAEAISDA AAEHDG CAS-L2 AAEYGAEA AAGAMFLE M10.007-stromelysin 3 (MMP-11) AAQNASMW A06.001-nodavirus endopeptidase AASGFASP M04.003-vibriolysin ADAHDG CAS-L3 ADAPKGGG
EKSAMEN I EMNE TBT4100 BIOKJEMI GRUNNKURS. 29. november 2007 kl
 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Professor Kjell M. Vårum, tlf. 93324 (mob. 930 22165) EKSAMEN
NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR BIOTEKNOLOGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Professor Kjell M. Vårum, tlf. 93324 (mob. 930 22165) EKSAMEN
NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE TFY4260 CELLEBIOLOGI OG CELLULÆR BIOFYSIKK
 Side av 1 av5 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE TFY4260 CELLEBIOLOGI OG CELLULÆR BIOFYSIKK Faglig kontakt under eksamen: Catharina Davies Tel 73593688 eller
Side av 1 av5 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK EKSAMEN I EMNE TFY4260 CELLEBIOLOGI OG CELLULÆR BIOFYSIKK Faglig kontakt under eksamen: Catharina Davies Tel 73593688 eller
Supplemental Data Regulation of MBK-2/DYRK by CDK-1 and the Pseudophosphatases EGG-4 and EGG-5 during the Oocyte-to-Embryo Transition
 Cell, Volume 139 Supplemental Data Regulation of MBK-2/DYRK by CDK-1 and the Pseudophosphatases EGG-4 and EGG-5 during the Oocyte-to-Embryo Transition Ken Chih-Chien Cheng, Richard Klancer, Andrew Singson,
Cell, Volume 139 Supplemental Data Regulation of MBK-2/DYRK by CDK-1 and the Pseudophosphatases EGG-4 and EGG-5 during the Oocyte-to-Embryo Transition Ken Chih-Chien Cheng, Richard Klancer, Andrew Singson,
4. Antigenpresentasjon til T celler. MHC molekyler.
 Immunologi 3. semester, V2015 4. Antigenpresentasjon til T celler. MHC molekyler. Karl Schenck Institutt for oral biologi TcR og MHC molekyler Noen hovedgrupper T celler som forlater thymus T celler med
Immunologi 3. semester, V2015 4. Antigenpresentasjon til T celler. MHC molekyler. Karl Schenck Institutt for oral biologi TcR og MHC molekyler Noen hovedgrupper T celler som forlater thymus T celler med
EKSAMEN I EMNE SIF4070 CELLEBIOLOGI Mandag 7. mai 2001 Tid: kl Ingen trykte eller håndskrevne hjelpemidler tillatt.
 Side av 5 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK Faglig kontakt under eksamen: Navn: Bjørn Torger Stokke Tlf: 93434 BOKMÅL EKSAMEN I EMNE SIF4070 CELLEBIOLOGI Mandag 7. mai
Side av 5 NORGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FOR FYSIKK Faglig kontakt under eksamen: Navn: Bjørn Torger Stokke Tlf: 93434 BOKMÅL EKSAMEN I EMNE SIF4070 CELLEBIOLOGI Mandag 7. mai
Cellebiologiske prosesser som respons på virusinfeksjon
 Cellebiologiske prosesser som respons på virusinfeksjon PBM 336 2005 Siri Mjaaland Infeksjoner - immunresponser 1 Figure 2-49 Interferoner Uspesifikk immunitet viral infeksjon stimulerer direkte produksjon
Cellebiologiske prosesser som respons på virusinfeksjon PBM 336 2005 Siri Mjaaland Infeksjoner - immunresponser 1 Figure 2-49 Interferoner Uspesifikk immunitet viral infeksjon stimulerer direkte produksjon
Løsningsforslag TFY 4260 Cellebiologi og cellulær biofysikk
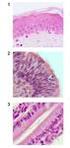 1 Løsningsforslag TFY 4260 Cellebiologi og cellulær biofysikk 18 mai 2013 Oppgave 1: Transport over epitelcellelag Fra tarm inn i epitelcellen med fasilitert diffusjon - Fasilitert diffusjon benytter bærerprotein
1 Løsningsforslag TFY 4260 Cellebiologi og cellulær biofysikk 18 mai 2013 Oppgave 1: Transport over epitelcellelag Fra tarm inn i epitelcellen med fasilitert diffusjon - Fasilitert diffusjon benytter bærerprotein
Frontiers in the Convergence of Bioscience and Information Technology
 Frontiers in the Convergence of Bioscience and Information Technology Frontiers in the Convergence of Bioscience and Information Technology Journal of Biomedicine and Biotechnology Frontiers in the Convergence
Frontiers in the Convergence of Bioscience and Information Technology Frontiers in the Convergence of Bioscience and Information Technology Journal of Biomedicine and Biotechnology Frontiers in the Convergence
IVA TEMPLE. Tools Enabling Metabolic Parents LEarning BASERT PÅ DEN ORIGINALE TEMPLE SKREVET AV BURGARD OG WENDEL VERSION 2, FEBRUAR 2017.
 TEMPLE Tools Enabling Metabolic Parents LEarning British Inherited Metabolic Diseases Group IVA BASERT PÅ DEN ORIGINALE TEMPLE SKREVET AV BURGARD OG WENDEL VERSION 2, FEBRUAR 2017 Støttet av IVA British
TEMPLE Tools Enabling Metabolic Parents LEarning British Inherited Metabolic Diseases Group IVA BASERT PÅ DEN ORIGINALE TEMPLE SKREVET AV BURGARD OG WENDEL VERSION 2, FEBRUAR 2017 Støttet av IVA British
FYS 3710 Biofysikk og Medisinsk Fysikk, DNA, RNA, Translasjon, Transkripsjon Proteinsyntese, Cellesyklus
 FYS 3710 Biofysikk og Medisinsk Fysikk, 2017 7 DNA, RNA, Translasjon, Transkripsjon Proteinsyntese, Cellesyklus Einar Sagstuen, Fysisk institutt, UiO 18.09.2017 1 DNA A / C / G / T 2 -deoxyribose monofosfate
FYS 3710 Biofysikk og Medisinsk Fysikk, 2017 7 DNA, RNA, Translasjon, Transkripsjon Proteinsyntese, Cellesyklus Einar Sagstuen, Fysisk institutt, UiO 18.09.2017 1 DNA A / C / G / T 2 -deoxyribose monofosfate
EKSAMEN I EMNE TBT4102 BIOKJEMI I. 10. desember 2010 kl
 NRGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FR BITEKNLGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Hanne Jørgensen, tlf. 591685 EKSAMEN I EMNE TBT4102 BIKJEMI I 10.
NRGES TEKNISK-NATURVITENSKAPELIGE UNIVERSITET INSTITUTT FR BITEKNLGI Faglig kontakt under eksamen: Institutt for bioteknologi, Gløshaugen Hanne Jørgensen, tlf. 591685 EKSAMEN I EMNE TBT4102 BIKJEMI I 10.
Hydrafacial Classic. Hydrafacial Classic
 Hydrafacial Classic Trinn 2: GlySal Prep 7,5% glykolsyre -2% salisylsyre Trinn 4: Helse for Huden Fjærlette strøk/tapping og hold 2 sekunder veil.pris inkl.mva: 1.495,- Hydrafacial Classic Activ-4 (melkesyre
Hydrafacial Classic Trinn 2: GlySal Prep 7,5% glykolsyre -2% salisylsyre Trinn 4: Helse for Huden Fjærlette strøk/tapping og hold 2 sekunder veil.pris inkl.mva: 1.495,- Hydrafacial Classic Activ-4 (melkesyre
Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
 Side 1 av 13 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
Side 1 av 13 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691), Janne Østvang (51278)
Fordøyelse i tidlige livsstadier:
 Proteinfordøyelse hos torsk Prosjektgruppe stor torsk: Grethe Rosenlund, Nutreco Ørjan Karlsen & Rolf Erik Olsen: HI Ann Cecilie Hansen & Gro-Ingunn Hemre, NIFES Torskelarver: Kristin Hamre, Audil Kvåle,
Proteinfordøyelse hos torsk Prosjektgruppe stor torsk: Grethe Rosenlund, Nutreco Ørjan Karlsen & Rolf Erik Olsen: HI Ann Cecilie Hansen & Gro-Ingunn Hemre, NIFES Torskelarver: Kristin Hamre, Audil Kvåle,
Biokjemiske reaksjonsruter. Glykolyse. Nedbrytning av glukose til cellulær energi
 Forelesning i glykolyse. Medisin stadium 1A, Geir Slupphaug, IKM Biokjemiske reaksjonsruter Kan virke svært forvirrende ved første øyekast. Slike oversikter er imidlertid ikke annet enn en form for kart,
Forelesning i glykolyse. Medisin stadium 1A, Geir Slupphaug, IKM Biokjemiske reaksjonsruter Kan virke svært forvirrende ved første øyekast. Slike oversikter er imidlertid ikke annet enn en form for kart,
Introduksjon til Biokjemi. Ingar Leiros, Institutt for Kjemi, UiT
 Introduksjon til Biokjemi Ingar Leiros, Institutt for Kjemi, UiT Biokjemi Biokjemi (Wikipedia): -Studien av de kjemiske prosesser i levende organismer, eller sagt på en annen måte; det molekylære grunnlaget
Introduksjon til Biokjemi Ingar Leiros, Institutt for Kjemi, UiT Biokjemi Biokjemi (Wikipedia): -Studien av de kjemiske prosesser i levende organismer, eller sagt på en annen måte; det molekylære grunnlaget
Søk. Nøkkelinformasjon. Sammendrag og figur. Klasser. IPC-klasse. Søker. Innehaver. Finn patenter, varemerker og design i Norge
 Søk Finn patenter, varemerker og design i Norge Nøkkelinformasjon Databasen er sist oppdatert Tittel Status Hovedstatus Detaljstatus Patentnummer Europeisk (EP) publiserings nummer 2017.11.04 12:03:00
Søk Finn patenter, varemerker og design i Norge Nøkkelinformasjon Databasen er sist oppdatert Tittel Status Hovedstatus Detaljstatus Patentnummer Europeisk (EP) publiserings nummer 2017.11.04 12:03:00
Hva er hovedoppgaven til kalsium ved aktivering av skjelett- (og hjerte-) muskulatur?
 Ved en konsentrisk muskelaksjon (muskelen forkortes) vil hver sarkomers A Avstand mellom Z linjene være uendret H og I bånd bli redusert og Z linjene kommer nærmere hverandre A bånd reduseres H bånd reduseres,
Ved en konsentrisk muskelaksjon (muskelen forkortes) vil hver sarkomers A Avstand mellom Z linjene være uendret H og I bånd bli redusert og Z linjene kommer nærmere hverandre A bånd reduseres H bånd reduseres,
Besvarelse eksamen i emnet TFY4260 Cellebiologi og cellulær biofysikk 1 juni 2010
 1 Besvarelse eksamen i emnet TFY4260 Cellebiologi og cellulær biofysikk 1 juni 2010 Oppgave 1: Transport over membraner. Na + /K + -ATPase pumpe Na+/K+ pumpen er et transmembranprotein som har 3 bindingssteder
1 Besvarelse eksamen i emnet TFY4260 Cellebiologi og cellulær biofysikk 1 juni 2010 Oppgave 1: Transport over membraner. Na + /K + -ATPase pumpe Na+/K+ pumpen er et transmembranprotein som har 3 bindingssteder
Faglig kontaktperson under eksamen: Jens Rohloff (mob 97608994)
 Side 1 av 6 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Jens Rohloff (mob 97608994) EKSAMEN I: BI1001
Side 1 av 6 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Jens Rohloff (mob 97608994) EKSAMEN I: BI1001
EKSAMEN I: BI1001 Celle- og molekylærbiologi BOKMÅL/ENGELSK VED SENSUR TELLER OPPGAVENE LIKT. VENNLIGST BESVAR HVER OPPGAVE PÅ NYTT ARK!
 Side 1 av Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691) EKSAMEN I: BI1001 Celle-
Side 1 av Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (98691) EKSAMEN I: BI1001 Celle-
ZINZINO. Helse konsept.
 ZINZINO Helse konsept www.izinzino.com De fleste ønsker å spise sunt, komme i form eller gå ned i vekt Den vanligste helseanbefalingen er å være i fysisk aktivitet. HVORFOR? Fordi det bygger muskler og
ZINZINO Helse konsept www.izinzino.com De fleste ønsker å spise sunt, komme i form eller gå ned i vekt Den vanligste helseanbefalingen er å være i fysisk aktivitet. HVORFOR? Fordi det bygger muskler og
Introduksjon til nevrotoksikologi
 Introduksjon til nevrotoksikologi Professor Tore Syversen Institutt for nevromedisin Det medisinske fakultet NTNU Tore.syversen@medisin.ntnu.no Aktualitet Metaller: kvikksølv, bly, aluminium, mangan Løsningsmidler
Introduksjon til nevrotoksikologi Professor Tore Syversen Institutt for nevromedisin Det medisinske fakultet NTNU Tore.syversen@medisin.ntnu.no Aktualitet Metaller: kvikksølv, bly, aluminium, mangan Løsningsmidler
Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (91897000) EKSAMEN I: BI1001 Celle- og molekylærbiologi BOKMÅL
 1 av 7 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (91897000) EKSAMEN I: BI1001 Celle-
1 av 7 Norges teknisknaturvitenskapelige universitet Fakultet for naturvitenskap og teknologi Institutt for biologi Faglig kontaktperson under eksamen: Berit Johansen (91897000) EKSAMEN I: BI1001 Celle-
